Documento sobre interpretación y utilidad clínica de los estudios de resistencia a antirretrovirales
Octubre 2023
Notas de la Versión:
FILIACIÓN Y CONFLICTO DE INTERESES
COORDINADORES:
José Luis Blanco Arévalo. Hospital Clinic de Barcelona. IDIBAPS. Universidad de Barcelona. CIBER de Enfermedades Infecciosas (CIBERINF)
JLB ha recibido compensación económica por asesorías, actividades formativas y ayudas a la investigación de Gilead Sciences, Janssen-Cilag, Theratechnology, Merck Sharp & Dohme y ViiV Healthcare.
Eva Poveda López. Instituto de Investigación Sanitaria Galicia Sur (IIS Galicia Sur), Hospital Álvaro Cunqueiro, Vigo.
EPL ha recibido ayudas para asistencia a reuniones científicas y honorarios por participación en asesorías científicas y actividades de educación biomédica por Gilead Sciences, ViiV Healthcare, Merck Sharp & Dohme, y TheraTechnologies.
REDACTORES:
Antonio Aguilera. Servicio y Departamento de Microbiología, Hospital Clínico Universitario de Santiago de Compostela y Universidade de Santiago de Compostela (USC). Instituto de Investigación Sanitaria de Santiago (IDIS).
AA ha recibido ayuda para asistencia a congresos y compensación económica por docencia y asesoría de Abbott, Abbvie, Diasorin, Gilead, Hologic, MSD, Roche y Siemens Healthcare.
Hortensia Alvarez. Unidad de Enfermedades Infecciosas. Complexo Hospitalario Universitario de Ferrol, A Coruña. HA ha recibido financiación para asistir y participar en congresos y reuniones científicas de Janssen-Cilag, Gilead Sciences y ViiV Healthcare.
Marta Alvarez. Servicio de Microbiología, Hospital Universitario Clínico San Cecilio, Granada. Instituto de Investigación BiosanitariaIbs. Granada.
José Luis Casado Osorio. Hospital Ramón y Cajal, Madrid
JCO ha recibido honorarios por ponencias para ViiV, Janssen, MSD y Gilead, así como honorarios por participación en comités asesores de Janssen y Gilead.
Rafael Delgado. Servicio de Microbiología. Hospital Universitario 12 de Octubre.
Instituto de Investigación Imas12
RD ha recibido compensación económica por participación en comités de asesoría para GSK y Gilead, Honorarios por conferencias Roche, MSD, Hologic, ViiV y Janssen.
Miguel García Deltoro, Servicio de Enfermedades Infecciosas. Consorcio Hospital General Universitario de Valencia.
MGD ha recibido honorarios por charlas, becas de investigación, patrocinio de cursos y participación en asesorías científicas: Gilead Sciences, ViiV Healthcare, Janssen, Merck Sharp & Dohme y Abbie, Menarini, Pfizer y Angelini.
Federico García García. Hospital Universitario Clínico San Cecilio, Ciber de Enfermedades Infecciosas CIBERINFEC, Instituto de Investigación BiosanitariaIbs.Granada.
FG ha recibido honorarios por charlas, becas de investigación, y participación en asesorías científicas: Gilead Sciences, ViiV Healthcare, Janssen, Merck Sharp & Dohme, TheraTechnologies, Abbvie, Werfen, Roche Diagn, Accelerate, Biomerieux, Hologic.
Africa Holguín. Instituto Ramón y Cajal para la Investigación Sanitaria (IRYCIS). Hospital Universitario Ramón y Cajal. Madrid. CIBER de Epidemiología y Salud Pública (CIBERESP).
AH no declara ningún conflicto de interés.
Arkaitz Imaz. Hospital Universitario de Bellvitge (L'Hospitalet de Llobregat, Barcelona)
AI ha recibido compensación económica por charlas y actividades formativas de Gilead Sciences, Janssen-Cilag, Merck Sharp & Dohme y ViiV Healthcare; por participación en consultorías, de Gilead Sciences, TheraTechnologies y ViiV Healthcare; y ayudas para la investigación de Gilead Sciences, Merck Sharp & Dohme, y ViiV Healthcare.
Ana María López Lirola. Servicio de Enfermedades Infecciosas. Hospital Universitario de Canarias. La Laguna. Santa Cruz de Tenerife.
ALL no declara ningún conflicto de interés.
Josep M Llibre. Servicio de Enfermedades Infecciosas, Hospital Universitari Germans Trias, Badalona.
JMLl ha recibido pagos por consultorías y actividades educacionales de ViiV Healthcare, Gilead Sciences, Thera Technologies y Janssen-Cilag. Es miembro del panel de expertos de las Guías de tratamiento antiretroviral de GeSIDA/PNS.
Carmen de Mendoza Fernández. Laboratorio de Medicina Interna. Hospital Puerta de Hierro-Majadahonda. IDIPHISA. Madrid.
CMF no declara ningún conflicto de interés.
Rocio Montejano. Unidad Enfermedades Infecciosas. Hospital Universitario La Paz
RM ha recibido honorarios por presentaciones, subvenciones a congresos y participación en asesorías científicas: Gilead Sciences, ViiV Healthcare, Merck Sharp & Dohme, Janssen-Cilag y Thera Technologies.
María del Mar Mosquera Gutiérrez. Servicio de Microbiología, Hospital Clínic de Barcelona. Universitat de Barcelona.
MMG no tiene conflictos de interés a declarar.
Roger Paredes. Servicio de Enfermedades Infecciosas. Hospital Universitari Germans Trias i Pujol
RP ha participado en advisory boards de Gilead, ViiV, GSK, Atea, MSD, Roche, Theratechnologies y Lilly y ha recibido financiación para estudios de ViiV y MSD.
María Jesús Pérez Elías. Hospital Ramón y Cajal, IRYCIS, CIBERINFECC.
MJPE ha recibido financiación para asistencia a congresos, actividades educacionales o de asesoría, así como becas de las compañías farmacéuticas Gilead, Janssen, Abbvie, MSD y ViiV-Healthcare.
Federico Pulido. Hospital Universitario 12 de Octubre, IMAS12, UCM. Madrid.
FP ha recibido honorarios por ponencias y asesoria de Gilead, Janssen, Merck y ViiV.
Carmen Ricart. Servicio de Enfermedades Infecciosas. Consorcio Hospital General Universitario de Valencia.
CR ha recibido becas investigación y patrocinio cursos Gilead Sciences, ViiV Healthcare, Janssen, Merck Sharp & Dohme y Abbvie. Patrocinio cursos también Menarini, Pfizer y Angelini.
Isabel Viciana Ramos. Servicio de Microbiología. UGC Infecciosos. Hospital Virgen de la Victoria. Málaga
IVR ha recibido becas investigación y patrocinio cursos de Gilead Sciences, ViiV Healthcare, Janssen, Merck Sharp & Dohme.
1. INTRODUCCIÓN
Autores: Eva Poveda y José Luis Blanco
Los estudios de resistencias a los fármacos empleados en el tratamiento de la infección por VIH son una herramienta fundamental para la correcta monitorización y seguimiento de los pacientes con esta infección. Sin embargo, las recomendaciones para su uso en la práctica clínica se han ido modificando y actualizando en función de los diferentes escenarios en cuanto a la prevalencia de mutaciones de resistencias, fármacos en uso, descripción de nuevas mutaciones y las metodologías disponibles. Desde el Grupo de Estudio del SIDA (GeSIDA) de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica (SEIMC) se presenta el Documento sobre la Utilidad Clínica de las Resistencias a Antirretrovirales que revisa los principales aspectos y recomendaciones a tener en cuenta para el uso de los estudios de resistencia en la práctica clínica actual.
El documento incluye 9 capítulos redactados por expertos nacionales en el estudio y manejo clínico de las resistencias a los antiretrovirales en los que se actualizan los aspectos más relevantes sobre la utilidad de los estudios de resistencias. Los capítulos describen de forma muy práctica las principales consideraciones a tener en cuenta en el contexto actual de la infección por VIH y nuestro entorno respecto a la situación epidemiológica sobre las resistencias, las recomendaciones nacionales para el tratamiento de la infección por VIH, la interpretación de las mutaciones a las diferentes familias de fármacos, las metodologías disponibles y sus limitaciones técnicas. Cada capítulo se acompaña de la bibliografía más relevante al respecto que permitirá profundizar y ampliar la información en caso necesario. Con todo ello, este documento pretende ser una guía práctica y actualizada para la realización de los estudios de resistencias en la práctica clínica teniendo en cuenta los últimos datos disponibles en este campo y la experiencia de los grandes profesionales en el tema.
2. SITUACIÓN EPIDEMIOLÓGICA DE LAS RESISTENCIAS A LOS ANTIRRETROVIRALES
Autores: Marta Álvarez y Carmen de Mendoza
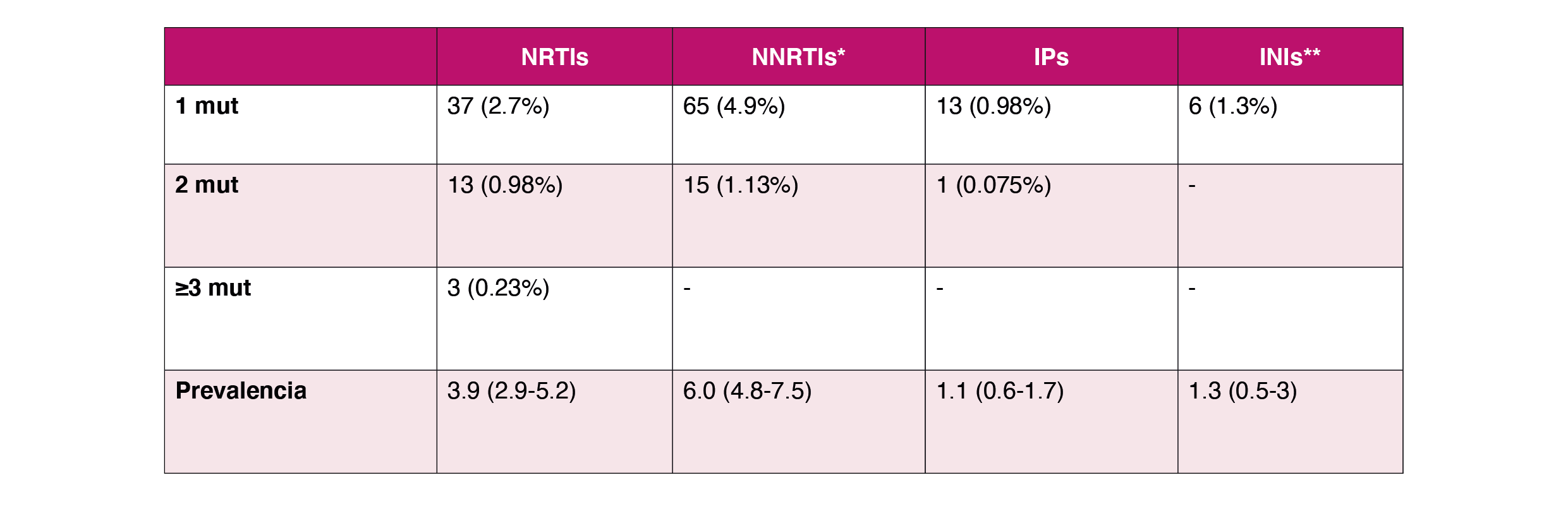
La Cohorte Española de pacientes naive, CoRIS, evalúa periódicamente la transmisión de variantes resistentes (TDR) en nuestro país. En el último análisis realizado para el período comprendido entre 2019-2020 el porcentaje global de mutaciones de resistencias fue del 11% (95% CI, 9.4%-13%) (Tabla 1)1 mientras que en el período 2007-2017 fue del 7.8% (95% CI, 7.1%-8.5%)2. Por familias de fármacos, las resistencias basales ITINAN son las más altas (6%), seguidas por los ITIAN (3.9%), mientras que para IPs se mantienen en un 1.1%. En cuanto a los INIs, la prevalencia se sitúa en un 1.3%, y en general, la transmisión de virus resistentes a esta familia continúa siendo baja. En la (Tabla 2) se recogen los cambios más frecuentemente observados en la cohorte española (CoRIS) distribuida por familia de antirretrovirales y periodo de estudio12.
El fracaso terapéutico con desarrollo de mutaciones de resistencias es a día de hoy excepcional y está en general limitado a casos anecdóticos34, incluso en pacientes con historia previa de resistencias45. Los factores que han modificado drásticamente la prevalencia de las resistencias en pacientes en situación de fracaso virológico son:
- Identificación del fracaso de forma precoz.
- La disponibilidad de un mayor arsenal terapéutico que facilita un diseño óptimo de terapias de rescate.
- El uso de combinaciones de tratamiento en una sola pastilla, una vez al día.
Las características de las resistencias en pacientes en situación de fracaso virológico a algunas combinaciones terapéuticas se resumen en los siguientes puntos:
- La aparición de mutaciones de resistencia en pacientes que fracasan a primera línea de tratamiento con esquemas a INIs es muy baja (<2%).
- De los inhibidores de la integrasa aprobados, RAL y EVG presentan una menor barrera a las resistencias y tienen un alto nivel de resistencias cruzadas, mientras que DTG, CAB y BIC presentan una barrera a las resistencias superior.
- El uso de terapias de mantenimiento/simplificación basadas en monoterapia con DTG se ha asociado con un mayor fracaso virológico con el consiguiente riesgo de aparición de mutaciones de resistencia y limitando el éxito de futuras terapias.
En aras de reducir toxicidad y favorecer la adherencia un elevado número de pacientes reciben combinaciones de tratamiento basadas en 2 antirretrovirales5. Es en estos supuestos en donde la vigilancia del desarrollo de resistencias puede tener un mayor interés. Hasta la fecha el desarrollo de resistencias en pacientes con fracaso virológico a combinaciones de 2 antirretrovirales es muy bajo (<1%). La (Tabla 3) resume las características del perfil de resistencias en pacientes con terapias de simplificación y mantenimiento 6 7 8 9 10 11 12 13
Tablas:
* WHO no incluye E138A
** Mutaciones detectadas en INIs no incluidas en IAS
2 Q95K** (0.5%), 5 T97A (1.16%), 3 A128T** (0.9%), 1 S153F (0.2%), 6 E157Q** (1.4%),
2 G163K** (0.5%), 2 D232N** (0.5%)


Bibliografía:
Viñuela L, Fuentes-López A, Serrano-Conde E, et al. Estudio de resistencias transmitidas en pacientes de nuevo diagnóstico en CoRIS: actualización 2019-20. XII Congreso Nacional GESIDA. 2021; CO-08.
Álvarez M, Fernández-Caballero JA, Pérez AB, et al. Decline in clinically relevant resistance to first line ARV regimens in Spain. 25th Conference on Retroviruses and Opportunistic Infections (CROI). 2018; Abstract 528.
Llibre JM, Viñuela L, de la Mora L, et al. Integrase resistance emergence with dolutegravir/lamivudine with prior HIV-1 suppression. J Antimicrob Chemother 2022; (online ahead of print)
Landman R, Marcelin AG, Bennani M, et al. COPEDOL: a two-year observational study in pretreated HIV-1 infected patients switching to a dolutegravir-based regimen. Infect Dis Now 2022; 52: 93-100
Pérez-Gonzalez A, Suarez-Garcia I, Ocampo A, Poveda E. Two-drug regimens for HIV- Current evidence, research gaps and future challenges. Microorganisms 2022; 10: 433
Rojas J, de Lazzari E, Negredo E, et al. Efficacy and safety of switching to dolutegravir plus lamivudine versus continuing triple antiretroviral therapy in virologically suppressed adults with HIV at 48 weeks (DOLAM): a randomized non-inferiority trial. Lancet HIV 2021; 8: e463-73
Osiyemi O, De Wit S, Ajana F, et al. Efficacy and safety of switching to dolutegravir/lamivudine versus continuing a tenofovir alafenamide-based 3- or 4-drug regimen for maintenance of virologic suppression in adults living with HIV-1: results through week 144 from the phase 3, non-inferiority TANGO randomized trial. Clin Infect Dis 2022; xx
Llibre JM, Brites C, Cheng C, et al. Efficacy and safety of switching to the 2-drug regimen dolutegravir/lamivudine versus continuing a 3- or 4- drug regimen for maintaining virological suppression in adults living with HIV-1: week 48 results from the phase 3, non-inferiority SALSA randomized trial. Clin Infect Dis 2022; XX
Deschanvres C, Reynes J, Lamaury I, et al. Dolutegravir-based dual maintenance regimens combines with lamivudine/emtricitabine or rilpivirine: risk of virological failure in a real-life setting. J AntimicrobChemother 2022; 77: 196-204.
Van Wyk J, Orkin C, Rubio R, et al. Durable suppression and low rate of virologic failure 3 years after switch to dolutegravir+rilpivirine 2-drug regimen: 148-week results from the sword-1 and sword-2 randomized clinical trials. J Acquir Immne Defic Syndr 2020; 85: 325-30
Maggiolo F, Gianotti N, Comi L, et al. Rilpivirine plus cobicistat-boosted darunavir as alternative to standard three-drug therapy in HIV-infected, virologically suppressed subjects: Final results of the PROBE 2 trial. Antivir Ther 2021; 26:51-7.
Orkin C, Bernal-Morell E, Tan D, et al. Initiation of long acting cabotegravir plus rilpivirine as direct to injection or with oral lead-in in adults with HIV-1 infection: week 124 results of the open-label phase 3 FLAIR study. Lancet HIV 2021; 8: e668-78.
Sutton K, De Vente J, Leblanc R, et al. Long-term efficacy, safety, and durability of cabotegravir and rilpivirine as 2-drug oral maintenance therapy after 6 year of study. Open Forum Infect Dis 2022; 9: ofac067.
3. RECOMENDACIONES SOBRE CUÁNDO HACER UNA PRUEBA DE RESISTENCIAS Y LOS ASPECTOS QUE HAY QUE CONSIDERAR
3.1. Aspectos a considerar para indicar un estudio de resistencias:
3.2. Limitaciones técnicas: carga viral mínima y especies minoritarias
3.3. Limitaciones clínicas
3.4. Indicación para solicitar un estudio de resistencias
Autores: José Luis Casado y Federico Pulido
Los estudios de resistencia (ER) a los antirretrovirales son una de las principales herramientas de las que dispone el clínico para optimizar el tratamiento antirretroviral1, pero como toda prueba de laboratorio es imprescindible conocer sus indicaciones, su interpretación y sus limitaciones.
Aspectos a considerar para indicar un estudio de resistencias:
Prevalencia de resistencias a los distintos antirretrovirales en nuestro medio
La prevalencia de mutaciones de resistencia transmitidas en poblaciones mayoritarias ha ido disminuyendo de forma paulatina en los últimos años en lo que se refiere a los ITIAN y a los IPs, frente a los ITINAN en los que ha aumentado discretamente2. En cuanto a los INIs, los datos de prevalencia de resistencias primarias en mutaciones claves es muy baja en el momento actual3.
Coste-eficacia de los test de resistencias
Conocer las resistencias basales es coste-eficaz si la prevalencia de resistencia en la población en estudio es superior al 5%, o incluso en cifras menores en caso de alto riesgo de fracaso4, aunque un reciente estudio no lo considera coste eficaz con el uso de fármacos con alta barrera a la resistencia5. En España, la tasa de resistencias transmitidas actual está en torno al 11%3: 6% para los ITINAN, 3,9% para los ITIAN, 1,1% para los IPs y 1,6% para los INIs.
Limitaciones técnicas: carga viral mínima y especies minoritarias
Carga viral mínima
Los ER utilizados en la clínica requieren un umbral de carga viral mínimo para garantizar la obtención de resultados. Aunque en los laboratorios con experiencia se consigue amplificar y secuenciar muestras por encima de 200 copias/mL5, la mayoría de los ensayos clínicos han adoptado un punto de corte de 400 copias/ml para realizar ER en caso de fracaso, con la suficiente reproducibilidad y menor variabilidad técnica. Sin embargo, actualmente con frecuencia los fracasos virológicos muestran valores de carga viral entre 50-400 copias/mL, es decir, en estos casos tendremos que valorar si esperar, con la posible evolución a mayor fracaso y resistencias78, o cambiar sin estar guiados por ER.
Especies minoritarias
Los ER convencionales (estudios de secuenciación poblacional o Sanger) no muestran poblaciones virales que estén presentes en menos del 15-20% de la carga viral total, por lo que no van a mostrar poblaciones minoritarias. De este modo, distintas mutaciones pueden permanecer ocultas a estos test de secuenciación de poblaciones virales mayoritarias y aparecer posteriormente en caso de reintroducción de fármacos a los que confieren resistencia9. En la actualidad existen diferentes plataformas que revisaremos en el capítulo 5 que pueden mostrar mutaciones que confieren resistencia y que están presentes en proporciones tan bajas como un 1%.
Limitaciones clínicas
Tiempo hasta el estudio de resistencias
Es importante saber si en el momento del estudio el paciente continúa con el tratamiento que ha fracasado, y si no es así cuánto tiempo ha pasado desde la interrupción del tratamiento. Se recomienda un plazo menor de 30 días entre la retirada y el estudio, pues el rápido recambio de la población viral conduciría a un predominio de poblaciones de virus salvaje que daría lugar a falsos negativos en la determinación de resistencias, o a una mezcla de subpoblaciones resistentes y salvaje con resultados contradictorios en el estudio genotípico de resistencias (mutaciones de resistencia) y fenotípico (sensible)10. Igualmente, se ha observado que el tiempo en fracaso, ajustado por el valor de la replicación viral y la barrera genética de los fármacos que se están recibiendo, puede determinar la tasa de resistencias. En una revisión de los fracasos con INIs (EVG) en ensayos clínicos pivotales, se observó que 4 de 18 pacientes no presentaban mutaciones de resistencia y solo se detectaban 51% de las mutaciones en la primera carga viral tras el fracaso, en comparación con la segunda carga viral de confirmación obtenida 4 a 6 semanas después11.
Dificultad en la interpretación del test de resistencias
Para interpretar un ER ante un fracaso virológico, es necesario conocer el tratamiento que estaba recibiendo el paciente en ese momento, y secuenciar todas las dianas terapéuticas implicadas (incluyendo la integrasa si el TAR incluía INIs). Se debe recordar que las mutaciones relacionadas con la resistencia a fármacos utilizados con anterioridad, pero que no están incluidos en el tratamiento actual, pueden no detectarse sin que ello signifique que no exista resistencia a dichos fármacos. Es por tanto conveniente: (i) utilizar todos los ER realizados en el seguimiento histórico del paciente a la hora de seleccionar el régimen de TAR que ofrezca las mayores garantías de eficacia, y (ii) considerar la posibilidad de la presencia de un virus resistente a aquellos fármacos con baja barrera a las resistencias si se confirma por historia un fracaso virológico en presencia de dichos fármacos (3TC/FTC, NEV, EFV, RPV e IPs no potenciados).
Indicación para solicitar un estudio de resistencias
En pacientes que nunca han recibido tratamiento antirretroviral:
La recomendación actual de tratar a todas las personas diagnosticadas de infección por VIH elimina la controversia del mejor momento para realizar un ER tras el diagnóstico1. Actualmente no se considera imprescindible disponer del ER a la hora de iniciar un TAR basado en INIs5 o IPs, y la realización de un ER no debería demorar el inicio del TAR, salvo en el caso de iniciar tratamiento con la combinación de DTG/3TC en individuos que se han infectado durante la toma de la PrEP dada la elevada prevalencia de la mutación M184V en este grupo de sujetos. En cualquier caso, se sigue recomendando la realización de ER basal, dada la relativa alta tasa de resistencias a ITINAN, y la posibilidad de uso futuro de ITIAN y ITINAN12. En el caso de no poder realizar ER basal, sería conveniente archivar una muestra de plasma previa al inicio de tratamiento para secuenciarla en el futuro si se plantea la utilización de un ITINAN.
Dada la casi nula tasa de resistencias transmitidas actuales a INIs, la secuenciación de la IN no está justificada1. La generalización del uso de INIs podría hacer cambiar la situación en el futuro, por lo que es imprescindible mantener un sistema de vigilancia epidemiológica que cuantifique la incidencia de resistencias transmitidas a INIs en nuestro entorno.
En el caso de una transmisión de fuente conocida con historia de fracaso al TAR, se debería realizar ER (con secuenciación de la integrasa en el caso de uso de este tipo de fármacos por el sujeto fuente).
En pacientes en fracaso virológico:
Ante un repunte virológico confirmado en un paciente con viremia previamente suprimida se debe investigar si ha habido: (i) una mala adherencia, o (ii) una interacción farmacocinética que haya reducido los niveles de los ARV. Si ha habido un abandono por un tiempo superior a 15-30 días el riesgo de falsos negativos del ER se incrementa, como hemos mencionado. En esa situación una alternativa sería –especialmente si la interrupción fue brusca y con fármacos con similar farmacocinética- reintroducir el TAR previo (si la interrupción no se ha debido a un problema de tolerancia) y reevaluar la necesidad del ER en función de la respuesta (como en el apartado anterior). Si no ha habido un abandono completo o éste es inferior a 15 días, la realización de un ER puede dar información sobre la posible selección de cepas resistentes a alguno o algunos de los fármacos utilizados en ese momento.
En mujeres embarazadas:
En el caso de una mujer embarazada en la que se va a iniciar TAR, siempre se debe realizar un ER, incluyendo la secuenciación de la integrasa si se va a prescribir un INI. El inicio de tratamiento no debe demorarse para esperar el resultado, pero disponer precozmente de la información en el caso de que existan resistencias transmitidas puede facilitar un ajuste rápido del TAR para conseguir el control virológico en el menor periodo de tiempo posible. Ante la presencia de una carga viral detectable durante el embarazo se recomienda realizar un ER para ajustar el TAR antes del parto.
Bibliografía:
Panel de expertos de Gesida y Plan Nacional sobre el Sida. Documento de consenso de Gesida/Plan Nacional sobre el Sida respecto al tratamiento antirretroviral en adultos infectados por el virus de la inmunodeficiencia humana. (Actualización 2022) Disponible en URL: https://gesida-seimc.org/wp-content/uploads/2022/05/GuiaGeSIDAPlanNacionalSobreElSidaRespectoAlTratamientoAntirretroviralEnAdultosInfectadosPorElVirusDeLaInmunodeficienciaHumanaActualizacionEnero2022.pdf
Álvarez M, Camacho-Luque R, Pérez AB, et al. Evolución en la transmisión de resistencias a antirretrovirales de primera línea en pacientes VIH en CoRIS (2007-2017). Programa de Vigilancia en la Integrasa Viral. IX Congreso Nacional de GeSIDA, 28 de noviembre – 1 de diciembre de 2017, Vigo. Abstract OR-05.
Viñuela-González L, Fuentes-López A, Serrano-Conde E, et al. Estudio de resistencias transmitidas en pacientes de nuevo diagnóstico en CoRIS: actualización 2019-20. XII Congreso Nacional de GeSIDA, 29 de noviembre – 2 de diciembre de 2021, Málaga. Abstract CO-08.
Sax P, Islam R, Walensky RP, et al. Should resistance testing be performed for treatment-naive HIV-infected patients? A cost-effectiveness analysis. Clin Infect Dis 2005; 41: 1316-23.
Koullias Y, Sax PE, Fields NF, et al. Should we be testing for baseline integrase resistance in patients newly diagnosed with human immunodeficiency virus?. Clin Infect Dis. 2017;65:1274-1281.
Stelzl E, Troppan KT, Winkler M, et al. Optimized protocol for detection of HIV-1 drug mutations in patients with low viral load. J Virol Methods. 2010; 168: 152–4.
Nettles RE, Kieffer TL, Simmons RP, et al. Genotypic resistance in HIV-1–infected patients with persistently detectable low-level viremia while receiving highly active antiretroviral therapy. Clin Infect Dis. 2004; 39: 1030–1037.
Swenson LC, Gonzalez-Serna A, Min JE, et al. HIV drug resistance occurring during low-level viremia is associated with subsequent virologic failure. Antivir Ther. 2013; 18: A1–A145.
Li JZ, Gallien S, Do TD, et al. Prevalence and significance of HIV-1 drug resistance mutations among patients on antiretroviral therapy with detectable low-level viremia. Antimicrob Agents Chemother.2012; 56: 5998–6000.
Taiwo, B. Understanding transmitted HIV resistance through the experience in the USA. Int J Infect Dis. 2009; 13: 552-9.
White K, Kulkami R, Miller MD. Analysis of early resistance development at the first failure timepoint in elvitegravir/cobicistat/emtricitabine/tenofovir disoproxil fumarate-treated patients. J Antimicrob Chemother.2015; 70: 2632-8.
Wittkop L, Gunthard HF, de Wolf F. Effect of transmitted drug resistance on virological and immunological response to initial combination antiretroviral therapy for HIV (EuroCoord-CHAIN joint project): a European multicohort study. Lancet Infect Dis. 2011; 11: 363–371.
Panel on Antiretroviral Guidelines for Adults and Adolescents. Guidelines for the use of antiretroviral agents in HIV-1 infected adults and adolescents. US Department of Health and Human Services. Disponible en URL: https://clinicalinfo.hiv.gov/sites/default/files/guidelines/documents/AdultandAdolescentGL.pdf. (Acceso 7 Marzo 2022).
4. RECOMENDACIONES TÉCNICAS EN LOS ESTUDIOS DE RESISTENCIAS
Autores: Antonio Aguilera y Rafael Delgado
Los estudios de resistencias tienen mayor validez si se realizan antes de iniciar, detener o cambiar el tratamiento. En la actualidad, están dirigidos frente a PR, TI, IN y envuelta (gp41 y V3). Sin embargo, otras regiones del genoma viral, como el gen gag, pueden tener importancia en la resistencia a otras familias como los IPs –aunque no se ha demostrado claramente- o a nuevas familias de antirretrovirales como los inhibidores de la maduración1. A continuación se presentan las principales recomendaciones técnicas a tener en cuenta para realizar un estudio de resistencia en la práctica clínica.
Tipo de muestra y almacenamiento
El plasma o el suero son las muestras de elección por su facilidad para procesar y almacenar. Además, la secuencia que vamos a obtener representa la cuasiespecie viral seleccionada más recientemente por la terapia antirretroviral2. En entornos con recursos limitados las muestras de sangre seca en papel de filtro (DBS) constituyen una alternativa3 a las muestras de plasma/suero. Los estudios en otros tipos de muestras como células mononucleares en sangre periférica (CMSP), semen o líquido cefalorraquídeo (LCR) no presentan en general problemas técnicos diferentes al plasma y constituyen un recurso que en algunas situaciones ha ayudado a entender la infección en diferentes compartimentos y a caracterizar variantes resistentes en territorios con baja penetración para algunos ARVs como el SNC4.
Las condiciones de almacenamiento (-80ºC para plasma) deben de ser las necesarias para mantener la integridad del ARN viral, aunque pueden variar en función de la muestra utilizada135.
Sensibilidad
Por razones técnicas, no se recomienda la realización de estudios de resistencia en pacientes en fracaso virológico con CV <500 cop/mL, ya que la probabilidad de amplificar el ARN viral es baja. En estos casos, se puede incrementar la sensibilidad mediante la concentración de un mayor volumen de muestra por ultracentrifugación1. En pacientes con CV indetectable o bajos niveles de viremia se suele recurrir a la realización de estudios de resistencia proviral en CMSP67, aunque no hay aún datos consistentes que avalen estos resultados.
Genotipo vs Fenotipo
Las pruebas genotípicas que detectan por secuenciación mutaciones conocidas asociadas a resistencia son ampliamente utilizadas por su simplicidad, rapidez, menor coste y buenos resultados a la hora del diseño del tratamiento de rescate1. Estas pruebas se realizan en la mayoría de los laboratorios de virología diagnóstica mediante procedimientos propios o alternativas comerciales: ViroSeq®-Abbott para secuenciación convencional (Sanger) o Sentosa®-Vela Diagnostics, ABL, Diagnóstica Longwood, etc para secuenciación masiva (NGS).
Durante los últimos años, la mayor parte de los estudios de resistencia a ARV se han realizado mediante técnicas genotípicas de secuenciación poblacional (Tecnología Sanger) en las que se obtiene una única secuencia representativa de todas las variantes circulantes presentes en la población viral. En la actualidad, la secuenciación masiva (deep sequencing o next-generation sequencing, NGS), que permite la detección de poblaciones virales minoritarias con una representación inferior al 15-20% en la población viral, se realiza en un mayor número de laboratorios y está progresivamente sustituyendo a la secuenciación poblacional8. En el capítulo 5 del documento se puede encontrar información detallada sobre la detección de poblaciones minoritarias. Una ventaja añadida de la utilización de NGS es la inclusión de las secuencias de PR, RT e IN sistemáticamente y en algunas técnicas la inclusión de V3 (tropismo).
Otros procedimientos genotípicos como la hibridación reversa, los microchips o la PCR-alelo específica ya no se encuentran disponibles comercialmente o se emplean de manera restringida en investigación.
En situaciones complejas con pacientes multitratados que acumulan un gran número de mutaciones y con escasas opciones terapéuticas, así como en la infección por VIH-2 o en ensayos clínicos con nuevos fármacos, las pruebas fenotípicas basadas en ensayos de virus recombinantes, en las que se estudia directamente el comportamiento del virus frente a compuestos específicos, podrían ser una opción8. Sin embargo, no están disponibles en la mayoría de los laboratorios y sus alternativas comerciales son igualmente limitadas (Phenosense® y Trofile®, Monogram BioScience).
Subtipos no-B
A pesar de que la sensibilidad a los ARVs y las mutaciones asociadas a resistencias son iguales para los subtipos B y no-B9, existen polimorfismos genéticos en los subtipos no-B que podrían modificar o facilitar la selección de resistencias. Desde un punto de vista técnico, esta variabilidad genética existente podría dificultar en algunos casos la determinación de los estudios de resistencia diseñados comercialmente2. En estos casos, se diseñarán cebadores específicos para conseguir resultados más óptimos (información más detallada en el capítulo 10).
Sistemas de interpretación
El creciente número de mutaciones asociadas a resistencia a los ARVs puede hacer muy compleja la interpretación de los resultados en los estudios genotípicos. Por esta razón, es recomendable la utilización de sistemas de interpretación automatizados para inferir el nivel de la resistencia a los ARV teniendo en cuenta todas las mutaciones detectadas en todos los estudios de resistencias que se ha realizado el paciente. Entre los numerosos sistemas disponibles uno de los más utilizados es el Stanford HIV Resistance Drug Database (HIVDB) de acceso libre y gratuito que ha incorporado también una versión para NGS. Existen otros sistemas de interpretación basados en reglas establecidas por expertos como el de la Red de Investigación en SIDA en España9, que incluyen también una App gratuita, disponible en Apple Store y Google Play, para dispositivos móviles y tablets (https://www.redris.es) En el documento encontrarán información detallada sobre la interpretación de la resistencias a las diferentes familias de fármacos (ver capítulos 6-9).
Estudios en ADN Proviral del VIH
Aunque no existen recomendaciones claras sobre su utilidad en la práctica clínica, la secuenciación del ADN proviral en CMSP es una alternativa para detectar mutaciones de resistencias archivadas y podría tener utilidad en pacientes con carga viral indetectable o pacientes con bajos niveles de viremia en los que los estudios sobre virus libre en plasma tiene un menor éxito de secuenciación del virus. Sin embargo, se han señalado discrepancias entre los resultados obtenidos en plasma y en células y su interpretación requiere especialmente de un experto en la interpretación de las resistencias1011.
Control de Calidad
Existen diferentes documentos16 que recogen recomendaciones con medidas referentes a establecer controles de calidad en los estudios de resistencia para evitar problemas de contaminación, así como errores en la identificación de las muestras. Estas medidas se basan fundamentalmente en la incorporación de distintos controles internos y externos y en los estudios filogenéticos de similitud.
Coste efectividad
Dadas las bajas tasas de transmisión de resistencias existentes descritas recientemente en nuestro país 12, actualmente está en discusión por su coste-efectividad la realización de estos estudios en pacientes sin tratamiento previo (ver capítulo 2 y 3).
Bibliografía:
Vandamme AM, Camacho RJ, Ceccherini-Silberstein F, et al. European recommendations for the clinical use of HIV drug resistance testing: 2011 update. AIDS reviews 2011; 13:77-108.
Shafer RW. Genotypic Testing for Human Immunodeficiency Virus Type 1 Drug Resistance. Clinical microbiology reviews 2002; 15:247-77.
Parkin N, de Mendoza C, Schuurman R, et al. Evaluation of in-house genotyping assay performance using dried blood spot specimens in the Global World Health Organization laboratory network. Clin Infect Dis 2012; 54 Suppl 4:S273-9.
Nightingale S, Geretti AM, Beloukas A, et al. Discordant CSF/plasma HIV-1 RNA in patients with unexplained low-level viraemia. Journal of neurovirology 2016; 22:852-60.
Garcia-Lerma JG, McNulty A, Jennings C, et al. Rapid decline in the efficiency of HIV drug resistance genotyping from dried blood spots (DBS) and dried plasma spots (DPS) stored at 37 degrees C and high humidity. J Antimicrob Chemother 2009; 64:33-6.
Poveda E, Paredes R, Moreno S, et al. Update on Clinical and Methodological Recommendations for Genotypic Determination of HIV tropism to Guide the Usage of CCR5 Antagonists. AIDS reviews 2012; 14:208-17.
Chew CB, Potter SJ, Wang B, et al. Assessment of drug resistance mutations in plasma and peripheral blood mononuclear cells at different plasma viral loads in patients receiving HAART. J Clin Virol 2005; 33:206-16.
Mbunkah HA, Bertagnolio S, Hamers RL et al. Low-Abundance Drug-Resistant HIV-1 Variants in Antiretroviral Drug-Naive Individuals: A Systematic Review of Detection Methods, Prevalence, and Clinical Impact. J Infect Dis 2020; 221: 1584–97.
Clutter DS, Jordan MR, Bertagnolio S, Shafer RW. HIV-1 drug resistance and resistance testing. Infect Genet Evol 2016; 46:292-307.
Delgado R. (Detection of resistance mutations in proviral DNA in HIV-1 infection). Enferm Infecc Microbiol Clin 2013; 31 Suppl 1:35-9.
Geretti AM, Blanco JL, Marcelin AG, et al. HIV DNA Sequencing to Detect Archived Antiretroviral Drug Resistance. Infectious Diseases and Therapy 2022.
Monge S, Diez M, Alvarez M, et al. Use of cohort data to estimate national prevalence of transmitted drug resistance to antiretroviral drugs in Spain (2007-2012). Clinical microbiology and infection : the official publication of the European Society of Clinical Microbiology and Infectious Diseases 2015; 21:105.e1-5.
5. RESISTENCIAS EN POBLACIONES MINORITARIAS: TÉCNICAS/PLATAFORMAS, INTERPRETACIÓN Y EVIDENCIAS
Autores: María del Mar Mosquera y Roger Paredes
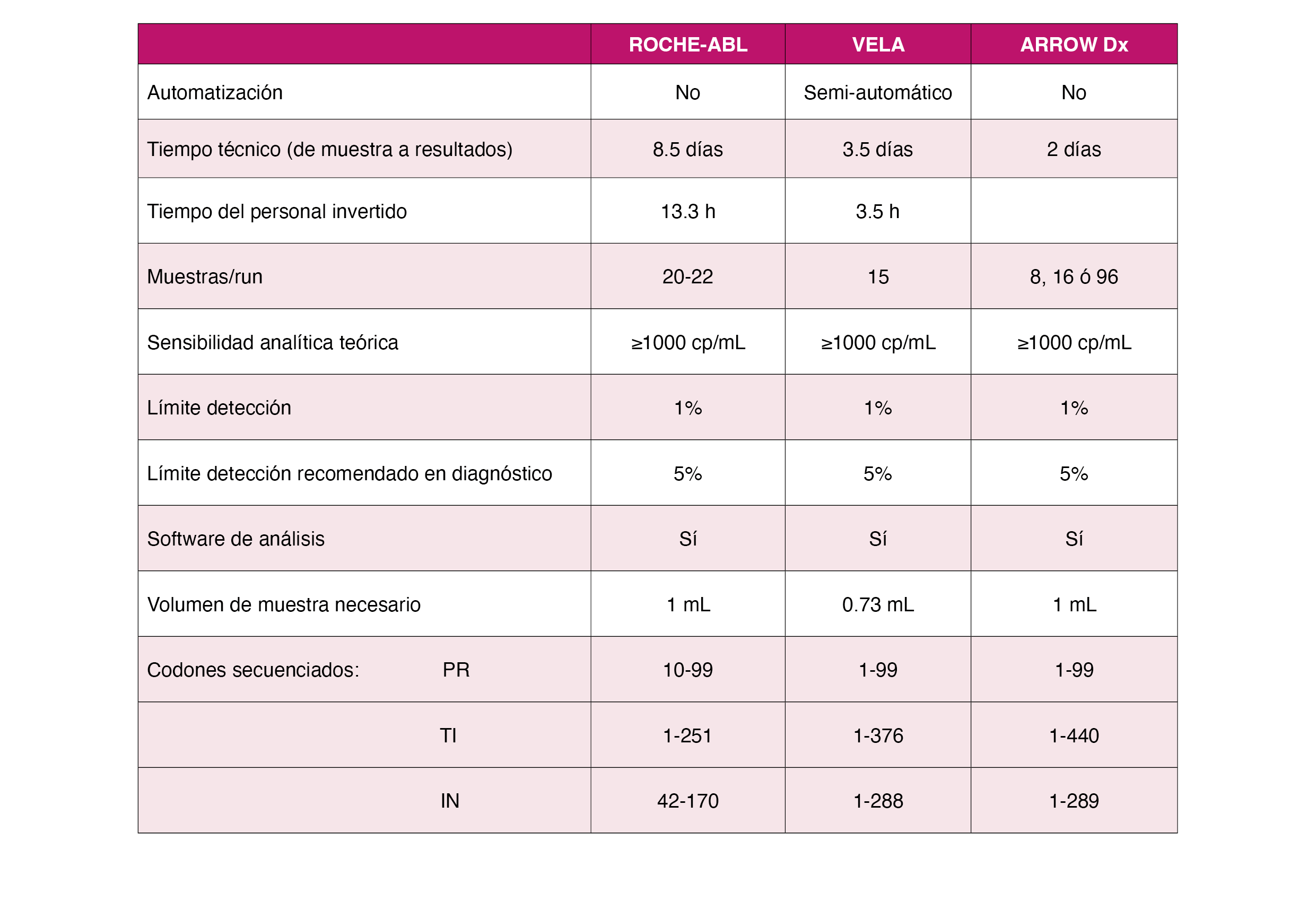
El genotipado viral utilizado tradicionalmente en diagnóstico clínico de la determinación de las mutaciones de resistencias se basa en la secuenciación por el método de Sanger. Este método únicamente es capaz de detectar mutantes virales aparezcan con una prevalencia mayor al 15-20% de la población viral. Con ello, mutantes resistentes presentes en poblaciones minoritarias, que en algunos casos podrían asociarse a fracaso del tratamiento, pasarían desapercibidas a la hora de diseñar un tratamiento efectivo. Mediante nuevas técnicas de secuenciación masiva, hoy es posible detectar mutantes presentes hasta en el 1-2% de la población viral de forma robusta y consistente. Dado que, además, dichas plataformas ofrecen la oportunidad de abaratar la secuenciación, distintos laboratorios han empezado a utilizarlas para el análisis de las resistencias genotípicas. Las plataformas de secuenciación masiva están en constante evolución1, pero muy pocas se están utilizando en la actualidad para diagnóstico clínico de VIH: MiSeqTM o ISeq 100TM (Illumina, San Diego, CA)23 y SentosaÒ SQ HIV Genotyping Assay (Vela Diagnostics, Singapur).
MiSeqTM es una plataforma abierta que se puede utilizar para múltiples aplicaciones de secuenciación. En 2016 Roche dejó de producir definitivamente el secuenciador GS Junior (454, Roche, Branford, CT) y desde entonces distribuye reactivos para la plataforma MiSeq (Illumina, San Diego, CA)23. ARROW Diagnostics S.R.L.(Genova, Italia) distribuye asimismo el kit AD4SEQ HIV-1 Solution v2 para su uso con los secuenciadores MiSeqTM y ISeq 100TM. Sentosa®, por su parte, es una plataforma basada en el método PGM Ion Torrent45 que recibió la marca CE-IVD en agosto de 2017 para genotipado de VIH Tabla 1. En estos sistemas, el análisis e interpretación de mutaciones se puede realizar mediante software accesible sin recurrir a complejos análisis bioinformáticos: DeepChekÒ-HIV (ABL, SA and TherapyEdge, Inc, Luxemburgo) en el caso de Roche-MiSeq, SmartVir (SmartSeq, http://www-smartseq.it/) en el caso de ARROW Diagnostica y Sentosa SQ Reporter en el caso de Vela. Además de estos softwares comerciales, distintos grupos han desarrollado plataformas web de análisis de resistencias automatizados, robustos y gratuitos, entre los que destacan PASeq (https://paseq.org, irsiCaixa) e Hydra (https://hydra.canada.ca, Public Health Agency of Canada)6.
Aunque tanto MiSeq como PGM Ion Torrent se han utilizado desde 2011 en investigación, los caminos hacia su comercialización como tests diagnósticos de rutina han sido distintos, y existen pocas comparaciones directas entre ellas. En términos generales, ambas plataformas producen resultados equivalentes. En una comparación realizada entre los sistemas Sentosa® y MiSeqTM la especificidad del primero en la detección de mutaciones resultó >99.9% en muestras con ARN de VIH-1 de al menos 500 copias/mL, presentando una reproducibilidad de cuantificación comparable a la de MiSeqTM7. Sin embargo, ambas plataformas utilizan una química de secuenciación distinta, lo que conlleva un patrón de errores diferente y utilizan software de corrección de errores distinto, por lo que es de esperar alguna discrepancia ocasional que, en términos generales, se espera que tenga poco impacto en la toma de decisiones clínicas. Se debe tener en cuenta que ambas plataformas están en constante proceso de mejora y actualización89. También está disponible comercialmente un ensayo de resistencia genotípica de secuenciación masiva que analiza ADN proviral de VIH-1 en células huésped: GenoSure Archive© (Monogram Biosciences) que genera secuencias de consenso para mutaciones presentes en ≥10% de las especies virales. Esta prueba puede proporcionar información sobre variantes virales resistentes que están archivadas dentro del ADN proviral. Sin embargo, pueden pasar por alto algunas o todas las mutaciones de resistencia previas que se han producido dentro de las cuasi-especies virales y, por lo tanto, deben interpretarse con precaución.
En la actualidad se pueden establecer las siguientes recomendaciones respecto a la utilización de métodos de secuenciación masiva en la práctica clínica:
1. Tanto las plataformas de secuenciación masiva como aquellas basadas en secuenciación Sanger son igualmente válidas para realizar estudios de resistencias genotípicas en el laboratorio clínico.
2. No existe una plataforma de secuenciación masiva mejor que otra. Se debe considerar que, en general, todas producen resultados equivalentes.
3. Los informes de estudios genotípicos de plataformas de secuenciación masiva siempre deben incluir los resultados a un nivel de sensibilidad Sanger-like, es decir, al 15 o 20% de sensibilidad (aunque las diferencias son mínimas, la mayoría de laboratorios están adoptando 15%). En general, los resultados a dicho nivel son los que deben guiar las decisiones clínicas.
- a) La única excepción a esta regla es si, por cualquier motivo, se considera iniciar tratamiento con NEV o EFV. Múltiples estudios demuestran que la detección de variantes minoritarias resistentes a estos fármacos se asocia a un mayor riesgo de fracaso virológico1011, por lo que en presencia de variantes minoritarias, dichos fármacos deberían evitarse. Algunos estudios sugieren que el límite de detección con una mayor exactitud diagnóstica sería el 5%12.
- b) No existen estudios suficientes que analicen el efecto de las variantes minoritarias en la respuesta a otros fármacos con barrera genética baja o intermedia, como los inhibidores de integrasa.
- c) La detección de variantes minoritarias no tiene ningún efecto sobre la efectividad de fármacos con barrera genética alta, como los inhibidores de la proteasa potenciados1314.
- d) La utilización de técnicas ultrasensibles puede mejorar la predicción del tropismo viral en comparación con los métodos de Sanger 15 16 17.
- e) Aunque no existe suficiente evidencia para dar recomendaciones definitivas, es posible que la detección de una mutante a una frecuencia entre 1 y 5% deba considerarse clínicamente relevante si se cumplen las dos condiciones siguientes:(i)Dicha mutación es capaz de conferir resistencia de nivel intermedio o alto al tratamiento por sí misma (ej. M184V a XTC, K65R a TFV, Q148R a RAL, etc.); (ii)Es congruente con la historia clínica del paciente, es decir, (a) ha sido detectada como variante mayoritaria anteriormente, o (b) se observa tras la exposición a un fármaco que es capaz de seleccionarla (ej. M184V o K65R tras profilaxis pre-exposición, Q148R en el contexto de fracaso virológico a inhibidores de integrasa).
- f)La detección de variantes minoritarias en otros compartimentos (LCR, semen, CMSP) debe enmarcarse en un contexto de investigación y no debe utilizarse para tomar decisiones clínicas. A menudo las variantes minoritarias en CMSP se producen en un contexto de edición por APOBECG en virus no viables. No existen estudios sólidos que demuestren su utilidad en el manejo del paciente.
Tablas:
Bibliografía:
Casadellà M, Paredes R. Deep sequencing for HIV-1 clinical management. Virus Res 2017; 239:69-81.
Bentley DR, Balasubramanian S, Swerdlow HP, et al. Accurate whole human genome sequencing using reversible terminator chemistry. Nature 2008; 456:53-9.
Ram D, Leshkowitz D, Gonzalez D, et al. Evaluation of GS Junior and MiSeq next-generation sequencing technologies as an alternative to Trugene population sequencing in the clinical HIV laboratory. J Virol Methods 2015; 212:12–6.
Chang MW, Oliveira G, Yuan J, Okulicz JF, Levy S, Torbett BE. Rapid deep sequencing of patient-derived HIV with ion semiconductor technology. J Virol Methods 2013; 189:232-4.
Rothberg JM, Hinz W, Rearick TM, et al. An integrated semiconductor device enabling non-optical genome sequencing. Nature 2011; 475:348-52.
Noguera-Julian M, Edgil D, Harrigan PR, Sandstrom P, Godfrey C, Paredes R. Next-Generation Human Immunodeficiency Virus Sequencing for Patient Management and Drug Resistance Surveillance. J Infect Dis 2017;216(suppl_9):S829-33.
Raymond S, Nicot F, Carcenac R, et al. HIV-1 genotypic resistance testing using the Vela automated next-generation sequencing platform. J Antimicrob Chemother 2018:1-6.
Brumme CJ, Poon AFY. Promises and pitfalls of Illumina sequencing for HIV resistance genotyping. Virus Res 2017; 239:97-105.
Loman NJ, Misra R V., Dallman TJ, et al. Performance comparison of benchtop high-throughput sequencing platforms. Nat Biotechnol 2012; 30:434-9.
Li JZ, Paredes R, Ribaudo HJ, et al. Low-Frequency HIV-1 Drug Resistance Mutations and Risk of NNRTI-Based Antiretroviral Treatment Failure: A Systematic Review and Pooled Analysis. JAMA [Internet] 2011;305(13):1327–35. Available from: papers://d08f9576-fa0b-499a-a277-114c394c193d/Paper/p3997.
Cozzi-Lepri A, Noguera-Julian M, Di Giallonardo F, et al. Low-frequency drug-resistant HIV-1 and risk of virological failure to first-line NNRTI-based ART: A multicohort European case-control study using centralized ultrasensitive 454 pyrosequencing. J Antimicrob Chemother 2015;70.
Ávila-Ríos S, García-morales C, Matías-florentino M, et al. Pretreatment HIV-drug resistance in Mexico and its impact on the eff ectiveness of fi rst-line antiretroviral therapy : a nationally representative 2015 WHO survey. Lancet HIV 2016; 3018:1-13.
Casadellà M, Manzardo C, Noguera-Julian M, et al. Clinical value of ultradeep HIV-1 genotyping and tropism testing in late presenters with advanced disease. Aids [Internet] 2015;29(12):1493–504. Available from: http://content.wkhealth.com/linkback/openurl?sid=WKPTLP:landingpage&an=00002030-201507310-00009.
Lataillade M, Chiarella J, Yang R, et al. Prevalence and clinical significance of HIV drug resistance mutations by ultra-deep sequencing in antiretroviral-naïve subjects in the CASTLE study. PLoS One 2010;5(6):e10952.
Swenson LC, Mo T, Dong WW, et al. Deep sequencing to infer HIV-1 co-receptor usage: application to three clinical trials of maraviroc in treatment-experienced patients. J Infect Dis [Internet] 2011;203(2):237-45. Available from: papers://d08f9576-fa0b-499a-a277-114c394c193d/Paper/p4172.
Swenson LC, Mo T, Dong WW, et al. Deep Third Variable Sequencing for HIV Type 1 Tropism in Treatment-Naive Patients: A Reanalysis of the MERIT Trial of Maraviroc. Clin Infect Dis [Internet] 2011;53(7):732-42. Available from: papers://d08f9576-fa0b-499a-a277-114c394c193d/Paper/p4178.
Mcgovern R, Thielen A, Mo T, et al. Population-based V3 genotypic tropism assay: a retrospective analysis using screening samples from the A4001029 and MOTIVATE studies. AIDS [Internet] 2010;24(16):2517-25. Available from: papers://d08f9576-fa0b-499a-a277-114c394c193d/Paper/p4177.
6. INTERPRETACIÓN DE RESISTENCIA A ITIAN
Autores: Isabel Viciana y Josep M Llibre
Los tratamientos antirretrovirales triples tanto de inicio como en simplificación suelen incluir una pareja coformulada de ITIAN compuesta por ABC/3TC o Tenofovir bien sea TDF o TAF combinado con FTC. ABC demostró una eficacia inferior en sujetos con CV>100.000 copias/mL cuando se administró con EFV o ATV/r.1 Esta diferencia vs tenofovir no se ha observado con INIs (RAL o DTG).234 Asimismo, los regímenes de tratamiento con dos fármacos incluyen siempre un inhibidor de la transcriptasa inversa, siendo lamivudina o rilpivirina los administrados con dolutegravir.
Hay 34 mutaciones no polimórficas en 15 posiciones en la transcriptasa inversa asociadas a resistencia a ITIAN,5 que se muestran en la Tabla 1. Los perfiles de mutaciones que afectan la actividad de estos análogos difieren ligeramente, por lo que es importante su análisis.
Los patrones de resistencias observados para TDF y TAF son muy similares, por lo que ambos fármacos serán referidos como “tenofovir”, aunque estudios in vitro demuestran pequeñas diferencias que favorecen a TAF en presencia de TAMs.678
Finalmente, diversos estudios confirman que la estimación de la actividad residual de los ITIAN en base a las mutaciones de resistencia es imperfecta. Así, en los estudios en TAR de segunda línea realizados en África, se obtienen tasas muy elevadas de respuesta con DRV/r o DTG combinados con 2 ITIAN en sujetos con fracaso virológico a una primera línea con ITINAN + 2 ITIAN, a pesar de tasas muy elevadas de resistencia tanto a 3TC como a tenofovir.9
Patrones de multiresistencia a ITIAN.
Existen básicamente tres vías de desarrollo de multiresistencia a ITIAN.
- Acúmulo de TAMs: M41L, D67N, K70R, L210W, T251Y/F, K219Q/E678. Clásicamente se agrupan en dos vías de desarrollo de resistencia: TAM 1 (M41L, L210W, T251Y) y TAM 2 (D67N, K70R, T215F, K219Q/E).101112 Confieren resistencia a todos los ITIAN con excepción de FTC o 3TC, la vía TAM 1 en mayor grado que TAM 2. En general, se considera que con ≥3 TAMs y especialmente con las mutaciones M41L y L210W, la resistencia es completa a todos los ITIAN excepto a tenofovir, que a menudo muestra resistencia intermedia y puede retener alguna actividad residual.1314 Tenofovir es el ITIAN que mantiene más actividad en presencia de TAMs. La mutación M184V revierte en algún grado la resistencia conferida por estas TAMs, especialmente en presencia de ≥3 TAMs.101315 El grado de resistencia cruzada depende del número y tipo de mutaciones y debe evaluarse en cada caso. TAF posee in vitro una mayor actividad antiviral que TDF frente al acúmulo de TAMs en diferentes aislados virales con diferentes patrones de resistencias.67 La presencia de TAMs causa un cierto grado de hipersusceptibilidad frente a ITINAN, aunque la traslación clínica de este hallazgo no está documentada. Las TAMs muestran un antagonismo fenotípico bidireccional con K65R y raramente suelen coexistir en el mismo genotipo.6781516
- Complejo de inserción de tres serinas entre los codones 67 y 70 (69SSS).10 Implica la inserción de ≥2 aminoácidos, más frecuentemente S-S, S-A, o S-G. Confiere resistencia a todos los ITIAN incluyendo tenofovir, especialmente cuando aparece junto a ≥1 TAM de la vía 1 (M41L, L210W o T251Y).
- Complejo de multiresistencia Q151M.10 Confiere resistencia a todos los ITIAN con excepción de tenofovir, salvo que esté presente la mutación K65R.(7,8,10,11,13-17) Incluye además las mutaciones A62V, V75I, F77L, F116Y. La mutación más relevante en este complejo es Q151M. El resto de mutaciones, de manera aislada, no confieren resistencia significativa.
M184V/I
Confiere resistencia fenotípica completa de alto grado a 3TC y FTC. Asimismo, confiere hipersusceptibilidad a AZT y TDF/TAF (en mayor grado in vitro a TAF).7812 Tiene un fuerte impacto en la capacidad replicativa o fitness viral y diversos ensayos clínicos han confirmado la eficacia clínica (sin añadir toxicidad) de mantener 3TC o FTC en un régimen antirretroviral de rescate a pesar de haberse confirmado la presencia de M184V, lo que se explicaría por la actividad residual que mantendría 3TC/FTC aún en presencia de la resistencia conferida por M184V.1819 Por sí sola confiere resistencia intermedia a ABC, y asociada a TAMs incrementa el grado de resistencia a ABC.
Diversos estudios demuestran que el porcentaje de M184V archivada tras fracasos previos se va reduciendo con el tiempo hasta hacerse a menudo indetectable si se mantiene una carga viral plasmática suprimida.20 Es probable que el impacto en un esquema de switch de una M184V archivada sea poco relevante si se ha mantenido una carga viral < 50 copias/mL durante más de 5 años.21
M184I se asocia a la presencia de E138K específicamente en fracasos a RPV, corrigiendo el defecto replicativo inducido por ésta. Aparecen probablemente debido a una neutralización incompleta de una o varias proteínas APOBEC3 que favorecen el escape viral.22 M184I suele emerger antes y de forma transicional a la M184V debido a que requiere una sustitución menos compleja de nucleótidos.
K65R
K65R es seleccionada principalmente por tenofovir, pero también, aunque con menor frecuencia, por ABC, d4T o ddI. La presencia de K65R está asociada con una respuesta virológica reducida a tenofovir. La existencia de TAMS o el tratamiento combinado con AZT previene la emergencia de K65R en presencia de tenofovir.716 No existen datos que indiquen diferencias en los patrones de resistencia entre TDF y TAF porque el componente activo de ambas formulaciones es tenofovir trifosfato.7814 Sin embargo TAF consigue niveles intracelulares entre 3 y 5 veces más elevados de tenofovir trifosfato que TDF.68 De hecho, TAF inhibió in vitro el crecimiento viral de 12/15 cepas con ≥3 TAMs, mientras que TDF (analizado in vitro a los niveles alcanzados con TDF) no fue capaz de inhibir el crecimiento de ninguna de las 15.68 La presencia de la mutación M184V/I aumenta la sensibilidad a tenofovir.(10-12,17) La presencia de M184V/I junto a K65R aumenta ligeramente la sensibilidad a tenofovir (que pasa de resistencia completa a resistencia intermedia de alto grado), AZT y d4T, y aumenta la resistencia a ddI y ABC.
K65R es seleccionada más frecuentemente (4-11%) en pacientes con subtipos no B en fracaso con d4T y también frente a tenofovir en subtipos C.232425
La selección de K65R en presencia de tenofovir se asocia a una disminución del fitness viral.17 La mutación K65R condiciona resistencia a tenofovir y resistencia intermedia a Abacavir, 3TC y FTC, y aumenta la sensibilidad a AZT.
K65E/N
La mutación K65N causa una ligera disminución de sensibilidad a tenofovir. K65E es una mutación raramente seleccionada por el tratamiento con ITIAN, que causa gran disminución de la fitness viral. 26 27
K70E/G/Q
La mutación K70E/G/Q es seleccionada por regímenes que contienen tenofovir o ABC. Están asociadas a un bajo nivel de resistencia a tenofovir, ABC y posiblemente a 3TC y FTC.514
L74V/I y Y115F
Son seleccionadas principalmente por ABC (también ddI), particularmente cuando los estudios de resistencias se realizan tras un prolongado tiempo de fracaso virológico. Y115F puede ser seleccionada también por tenofovir.517 Y115F causa resistencia a ABC y bajo nivel de resistencia a tenofovir. L74V/I causa resistencia intermedia a ABC (y elevada a ddI) y cierta hipersusceptibilidad a tenofovir.
Tablas:
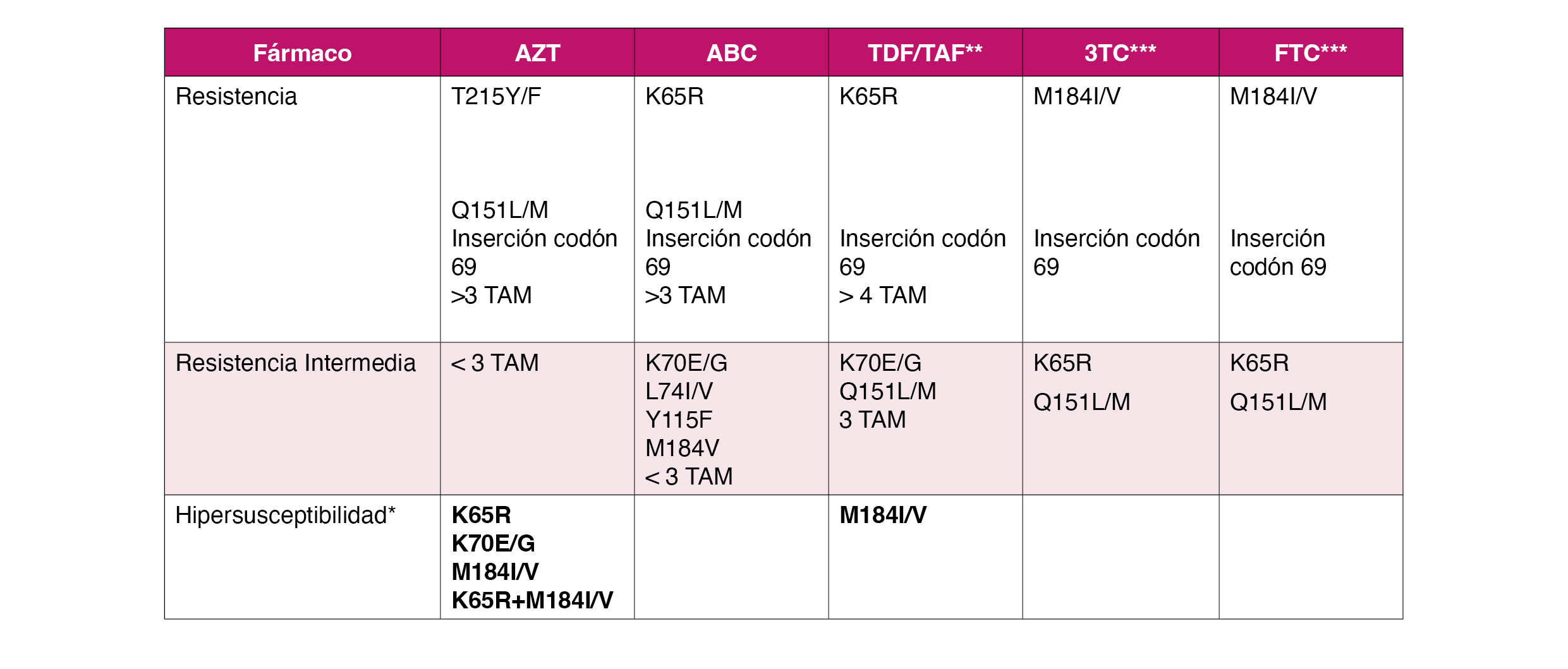
AZT: Zidovudina; ABC: Abacavir; TDF/TAF: Tenofovir/ Tenofovir Alafenamida; 3TC: Lamivudina; FTC: Emtricitabina
*La presencia de mutaciones que confieren hipersusceptibilidad (verde) junto a mutaciones que confieren resistencia tendrían una interpretación de Resistencia Intermedia, con la excepción de la K65R+M184I/V que revertiría la resistencia al AZT.
**A pesar de que TDF y TAF tienen algunas diferencias in vitro en sus CI50 y actividad frente a algunas mutaciones, este comité recomienda la misma valoración para ambos en base a los resultados de los ensayos clínicos.
***En caso de historia de fracaso previo a un TAR que contenga 3TC ó FTC debe de considerarse resistencia a estos fármacos.
Bibliografía:
Sax PE, Tierney C, Collier A, FIschl M. Abacavir–Lamivudine versus Tenofovir– Emtricitabine for Initial HIV-1 Therapy. N Engl J Med 2009;361:2230–40.
Raffi F, Rachlis A, Stellbrink HJ, et al. Once-daily dolutegravir versus raltegravir in antiretroviral-naive adults with HIV-1 infection: 48 week results from the randomised, double-blind, non-inferiority SPRING-2 study. Lancet 2013;381(9868):735–43.
Clotet B, Feinberg J, Van Lunzen J, et al. Once-daily dolutegravir versus darunavir plus ritonavir in antiretroviral-naive adults with HIV-1 infection (FLAMINGO): 48 week results from the randomised open-label phase 3b study. Lancet 2014;383(9936):2222–31.
Gallant J, Lazzarin A, Mills A, et al. Bictegravir, emtricitabine, and tenofovir alafenamide versus dolutegravir, abacavir, and lamivudine for initial treatment of HIV-1 infection (GS-US-380-1489): a double-blind, multicenter, phase 3, randomised controlled non-inferiority trial. Lancet 2017;390(10107):2063–72.
Bennett DE, Camacho RJ, Otelea D, et al. Drug resistance mutations for surveillance of transmitted HIV-1 drug-resistance: 2009 update. PLoS One 2009;4(3).
Margot NA, Liu Y, Miller MD, Callebaut C. High resistance barrier to tenofovir alafenamide is driven by higher loading of tenofovir diphosphate into target cells compared to tenofovir disoproxil fumarate. Antiviral Res 2016;132:50–8.
Margot NA, Johnson A, Miller MD, Callebaut C. Characterization of HIV-1 resistance to tenofovir alafenamide in vitro. Antimicrob Agents Chemother 2015;59(10):5917–24.
Margot N, Ram R, Abram M, Haubrich R, Callebaut C. Antiviral Activity of Tenofovir Alafenamide against HIV-1 with Thymidine Analog-Associated Mutations and M184V. Antimicrob Agents Chemother 2020;64(4):1–11.
Paton NI, Musaazi J, Kityo C, et al. Dolutegravir or Darunavir in Combination with Zidovudine or Tenofovir to Treat HIV. N Engl J Med 2021;385(4):330–41.
Whitcomb JM, Parkin NT, Chappey C, Hellmann NS, Petropoulos CJ. Broad Nucleoside Reverse‐Transcriptase Inhibitor Cross‐Resistance in Human Immunodeficiency Virus Type 1 Clinical Isolates. J Infect Dis 2003;188(7):992–1000.
Melikian GL, Rhee SY, Taylor J, et al. Standardized comparison of the relative impacts of HIV-1 Reverse Transcriptase (RT) mutations on nucleoside RT inhibitor susceptibility. Antimicrob Agents Chemother 2012;56(5):2305–13.
Miller MD, Margot N, Lu B, et al. Genotypic and phenotypic predictors of the magnitude of response to tenofovir disoproxil fumarate treatment in antiretroviral-experienced patients. J Infect Dis 2004;189(5):837–46.
Wolf K, Walter H, Beerenwinkel N, et al. Tenofovir Resistance and Resensitization. Antimicrob Agents Chemother 2003;47(11):3478–84.
Wensing AM, Calvez V, Ceccherini-Silberstein F, et al. 2019 update of the drug resistance mutations in HIV-1. Top Antivir Med 2019;27(3):111–21.
Parikh UM, Bacheler L, Koontz D, Mellors JW. The K65R Mutation in Human Immunodeficiency Virus Type 1 Reverse Transcriptase Exhibits Bidirectional Phenotypic Antagonism with Thymidine Analog Mutations. J Virol 2006;80(10):4971–7.
Von Wyl V, Yerly S, Böni J, et al. Factors associated with the emergence of K65R in patients with HIV-1 infection treated with combination antiretroviral therapy containing tenofovir. Clin Infect Dis 2008;46(8):1299–309.
White KL, Margot NA, Wrin T, Petropoulos CJ, Miller MD, Naeger LK. Molecular mechanisms of resistance to human immunodeficiency virus type 1 with reverse transcriptase mutations K65R and K65R+M184V and their effects on enzyme function and viral replication capacity. Antimicrob Agents Chemother 2002;46(11):3437–46.
Paton NI, Kityo C, Hoppe A, et al. Assessment of Second-Line Antiretroviral Regimens for HIV Therapy in Africa. N Engl J Med 2014;371(3):234–47.
Campbell TB, Shulman NS, Johnson SC, et al. Antiviral activity of lamivudine in salvage therapy for multidrug-resistant HIV-1 infection. Clin Infect Dis 2005;41(2):236–42.
Palich R, Teyssou E, Sayon S, et al. Kinetics of archived M184V mutation in treatment-experienced virally suppressed HIV-infected patients. J Infect Dis 2021;225(3):502–9.
Santoro M, Armenia D, Teyssou E, Santos J, Charpentier C, Lambert-Niclot S. The impact of M184V on the virological efficacy of switch to 3TC/DTG in real life. Conf Retroviruses Opportunistic Infect virtual March 6-11, 2021; #429
Fourati S, Malet I, Lambert S, et al. E138K and M184I mutations in HIV-1 reverse transcriptase coemerge as a result of APOBEC3 editing in the absence of drug exposure. Aids 2012;26(13):1619–24.
Hawkins CA, Chaplin B, Idoko J, et al. Clinical and genotypic findings in HIV-infected patients with the k65r mutation failing first-line antiretroviral therapy in Nigeria. J Acquir Immune Defic Syndr 2009;52(2):228–34.
Gregson J, Kaleebu P, Marconi VC, et al. Occult drug resistance to thymidine analogues and multidrug resistant HIV-1 following failure of first line tenofovir-based antiretroviral regimens in sub Saharan Africa: a retrospective multicentre cohort study. Lancet Infect Dis 2016;
Skhosana L, Steegen K, Bronze M, et al. High prevalence of the K65R mutation in HIV-1 subtype C infected patients failing tenofovir-based first-line regimens in South Africa. PLoS One 2015;10(2):1–12.
Fourati S, Visseaux B, Armenia D, et al. Identification of a rare mutation at reverse transcriptase Lys65 (K65E) in HIV-1-infected patients failing on nucleos(t)ide reverse transcriptase inhibitors. J Antimicrob Chemother 2013;68(10):2199–204.
Chunduri HB, Crumpacker C, Sharma PL. Reverse transcriptase mutation K65N confers a decreased replication capacity to HIV-1 in comparison to K65R due to a decreased RT processivity. Virology 2011;414(1):34–41.
7. INTERPRETACIÓN DE RESISTENCIA A ITINAN
Autores: María Jesús Pérez-Elias y Arkaitz Imaz
Actualmente están comercializados 5 ITINAN: NVP, EFV, ETR, RPV y DOR1. Ninguno de ellos está indicado como tratamiento de inicio preferente en la mayor parte de las guías de tratamiento antirretroviral del adulto1. El uso de los ITINAN de primera generación NVP y EFV es cada vez menor. DOR y RPV están recomendados como pautas alternativas en pacientes naive –aunque pueden ser de elección en algunos casos–y como en cambios de tratamiento en pacientes con supresión virológica. La mayor evidencia para el uso de ETR es en pautas de rescate avanzado1
Los ITINAN son moléculas pequeñas que inhiben la TI fijándose a un bolsillo hidrofóbico de la enzima en una unión alostérica, modificando su flexibilidad y bloqueando la síntesis de ADN. La secuencia de la TI que codifica el bolsillo de unión de los ITINAN comprende la secuencia de aminoácidos entre las posiciones 90 y 348. Las mutaciones que comprometen más la actividad de los ITINAN están ubicadas fundamentalmente en las regiones 98-108 y 179-190 de la TI, aunque también pueden ocurrir entre los codones 225 y 2382. Se han encontrado MR en los codones 90, 98, 100, 101, 103, 106, 108, 138, 179, 181,188, 190, 221, 225, 227, 230, 236, 238, 318, 348.
La primera MR que aparece tras el fracaso virológico con una pauta basada en ITINAN, depende del fármaco al que se ha expuesto el paciente (normalmente EFV: K103N; NVP: Y181C; y ETR y RPV: E138K; DOR: V106A y F227C).
La resistencia cruzada entre ellos es muy frecuente y especialmente entre los de primera generación EFV y NVP3.
La mutación K103N, la más frecuente en pacientes con fracaso previo con EFV, no tiene impacto por si sola en la resistencia a los ITINAN de segunda generación ETR, RPV o DOR.
ETR tiene una mayor barrera a las resistencias y se requiere la acumulación de varias mutaciones para condicionar resistencia de alto nivel. Es el único ITIANN en el que las mutaciones están ponderadas por su impacto en la resistencia que confieren.
RPV tiene un perfil de resistencias diferenciado de los ITINAN de primera generación, con similitudes a ETR, pero su barrera genética es baja y una sola mutación puede ser suficiente para conferir resistencia de alto nivel. Las mutaciones K101E, E138K, y Y181C confieren resistencia a RPV y son las que, en general, aparecen primero en pacientes con fracaso a una pauta con RPV. Cuando la mutación E138K o K101E aparecen en combinación con la mutación M184V de resistencia a ITIAN, se incrementa el nivel de resistencia a RPV4.
La tasa de fracaso virológico, observada en los ensayos clínicos con la combinación de CAB/RPV de acción prolongada, en el escenario de cambio con CV suprimida, ha sido baja (1%). Las mutaciones a RPV archivadas en DNA proviral, en combinación con al menos otro factor, el subtipo viral A1/A6 con el polimorfismo L74I, o un IMC>30Kg/m2, se ha asociado con mayor tasa de fracaso a esta pauta de TAR. Por tanto, es importante tener en cuenta si existen mutaciones históricas que confieren resistencia a RPV o la posibilidad de haberlas adquirido durante un fracaso previo con ITINAN5. En un estudio francés sobre 4212 pacientes naive, se encontró que la presencia de al menos una MR a CAB o RPV fue del 16,2 % y del 14,3 %, respectivamente. Utilizando el algoritmo del ANRS, el 0,74 % (n = 31), el 6,2 % (n = 261) y solo el 0,09 % (n = 4) de las secuencias fueron resistentes a CAB o RPV o ambos, respectivamente. La prevalencia de L74I en la integrasa y E138A en la RT fue del 13,0 % y el 3,2 %, respectivamente. Se encontraron 183 secuencias del subtipo A6/A1 y 244 secuencias interpretadas como resistentes a rilpivirina, en 427 (10,1 %), pero la combinación de ambos fue mucho menos frecuente 0,4%6.
DOR tiene un perfil de resistencias parcialmente diferenciado del resto de ITINAN, manteniendo actividad en presencia de mutaciones como K103N, E138K, Y181C o G190A. En general se requiere más de una mutación para conferir resistencia de alto nivel a DOR. Individualmente, las mutaciones con mayor impacto para DOR son V106A, Y188L, y M230L.
A diferencia de los ITINAN de primera generación, la resistencia primaria a DOR en pacientes naive en nuestro medio es muy poco prevalente (<1%)78. En pacientes pretratados, el uso previo de EFV o ETR se ha asociado con mayor probabilidad de resistencia a DOR y el antecedente de tratamiento con RPV con una menor probabilidad de resistencia a DOR9.
Una característica común de los ITINAN es su falta de actividad virológica residual en presencia de MR que les afectan10.
Mutaciones de Resistencia en pacientes Naive
En los últimos registros epidemiológicos a nivel mundial, se ha observado un aumento considerable de las MR asociadas a los ITINAN, especialmente en países de África Subsahariana, Latinoamérica y Asia11 En España los ITINAN son la familia con mayor prevalencia de MR en pacientes naive12. Esto tiene poca relevancia en nuestro país, al no ser fármacos de uso preferente en el TAR inicial1. Sin embargo, merece consideración cuando se plantee modificar el TAR en pacientes con supresión virológica. En la lista actualizada de MR para la vigilancia epidemiológica de las resistencias trasmitidas a ITINAN se incluyen 19 MR, en 10 posiciones de la TI: 100I; 101E/P; 103N/S; 106M/A; 179F; 181C/I/V; 188L/H/C; 190A/S/E; 225H; 230L13.
Las MR K103N, Y181C y G190A son las encontradas en más del 80% de cepas con MR transmitidas a ITINAN de primera generación, en cualquier área geográfica y subtipo viral. V106M es la cuarta MR más frecuente en el subtipo C14.
En la práctica clínica los pacientes con MR aisladas a los ITINAN pueden recibir en primera línea, un régimen basado en INI o en un IP15.
Interpretación de las Mutaciones de Resistencia
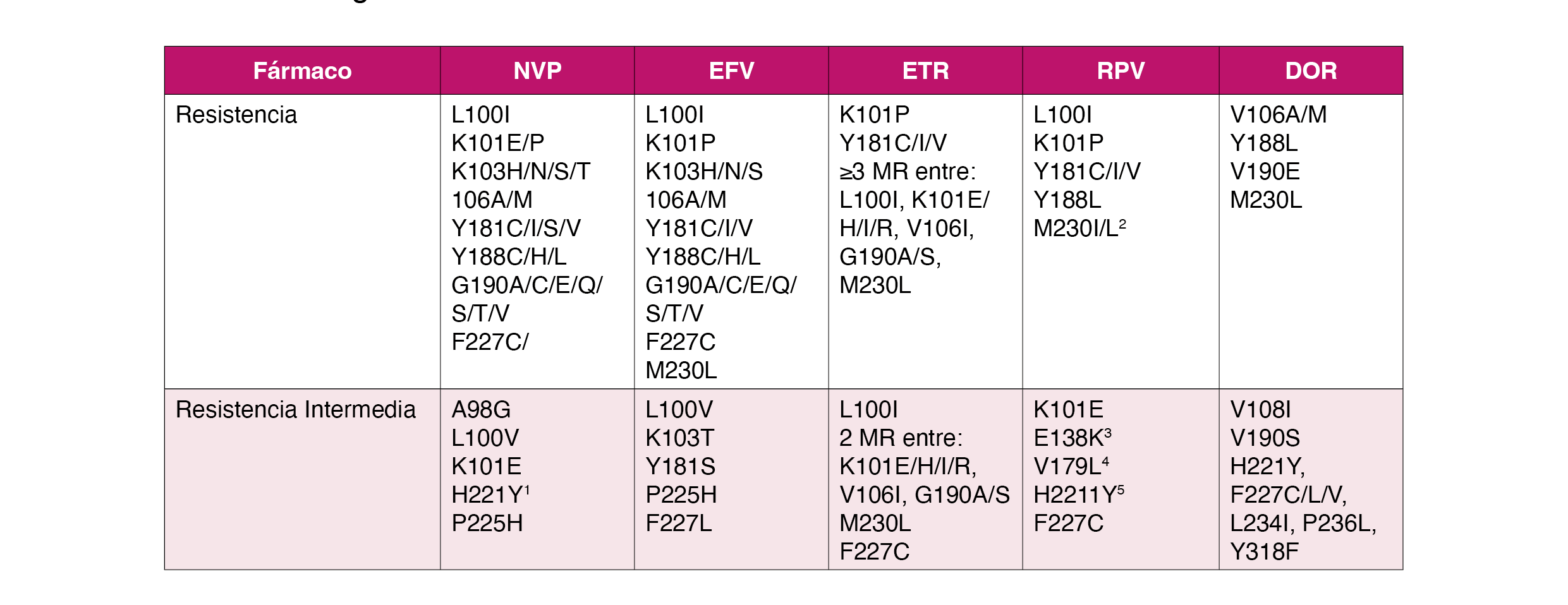
En la Tabla 1 se muestra la interpretación de las diferentes mutaciones en la sensibilidad de los diferentes ITINAN161718 En algunos subtipos se han descrito mutaciones particulares, A98S en el subtipo C19 o L228I y Y232H en el subtipo CRF08_BC20.
Tablas:
Bibliografía:
Documento de consenso de GeSIDA/ División de Control de VIH, ITS, Hepatitis virales y Tuberculosis del Ministerio de Sanidad respecto al tratamiento antirretroviral en adultos infectados por el virus de la inmunodeficiencia humana (Actualización enero 2023) en http://gesida-seimc.org/category/guias-clinicas/antirretroviral-vigentes/
Wei X, Ghosh SK, Taylor ME, et al. Viral dynamics in human immunodeficiency virus type 1 infection. Nature 1995; 373:117-22.
Delaugerre C, Rohban R, Simon A, et al. Resistance profile and cross-resistance of HIV-1 among patients failing a non-nucleoside reverse transcriptase inhibitor-containing regimen. J Med Virol 2001; 65:445-48.
Vingerhoets J, Tambuyzer L; Azijn H, et al. Resistance profile of etravirine: combined analysis of baseline genotypic and phenotypic data from the randomized, controlled Phase III clinical studies. AIDS 2010; 24:503-14.
Cutrell AG, Schapiro JM, Perno CF, et al. Exploring predictors of HIV-1 virologic failure to long-acting cabotegravir and rilpivirine: a multivariable analysis. AIDS. 2021 Jul 15;35(9):1333-1342.
Charpentier C, Storto A, Soulié C, et al. Prevalence of genotypic baseline risk factors for cabotegravir + rilpivirine failure among ARV-naive patients. J Antimicrob Chemother. 2021 Oct 11;76(11):2983-2987. doi: 10.1093/jac/dkab161. Erratum in: J Antimicrob Chemother. 2021 Sep 17
Soulie C, Santoro MM, Charpentier C, et al. Rare occurrence of doravirine resistance-associated mutations in HIV-1-infected treatment-naive patients. J Antimicrob Chemother. 2019 Mar 1;74(3):614-617. doi: 10.1093/jac/dky464. PMID: 30476106.
Guerrero-Beltrán C, Martínez-Sanz J, Álvarez M, et al. The algorithm used for theinterpretation of doravirine transmitted drug resistance strongly influences
clinical practice and guideline recommendations. J Antimicrob Chemother. 2020 May 1;75(5):1294-13 0. doi: 10.1093/jac/dkaa009. PMID: 32030406.
Sterrantino G, Borghi V, Callegaro AP, et al. Prevalence of predictedresistance to doravirine in HIV-1-positive patients after exposure to non-
nucleoside reverse transcriptase inhibitors. Int J Antimicrob Agents. 2019 Apr;53(4):515-519. doi: 10.1016/j.ijantimicag.2019.02.007. Epub 2019 Feb 12.
PMID: 30769200.
Deeks S, Hoh R, Torsten B. et al. Interruption of Treatment with Individual Therapeutic Drug Classes in Adults with Multidrug‐Resistant HIV‐1 Infection. J Infect Dis 2005; 192:1537–44.
Gupta R, Gregson J, ParkinN, et al. HIV-1 drug resistance before initiation or re-initiation of first-line antiretroviral therapy in low-income and middle-income countries: a systematic review and meta-regression analysis. Lancet Infect Dis 2017; 8: 346–55
Alvarez M, Fernández-Caballero JA, Perez A, et al. Decline in clinically relevant resistance to first line ARV regimens in Spain. XXV Conference on Retroviruses and Opportunistic Infections (CROI) Boston, Massachusetts, March 4-7, 2018. Abstract 528.
Bennett DE, Camacho RJ, Otelea D, et al. Drug Resistance Mutations for Surveillance of Transmitted HIV-1 Drug-Resistance: 2009 Update. PLOS ONE 2009; 4: e4724. https://doi.org/10.1371/journal.pone.0004724
Rhee SY, Blanco JL, Jordan MR, et al. Geographic and temporal trends in the molecular epidemiology and genetic mechanisms of transmitted HIV-1 drug resistance: an individual-patient- and sequence-level meta-analysis.PLoS Med. 2015 Apr 7;12(4): e1001810.
Clutter DS, Fessel WJ, Rhee SY, et al. Response to Therapy in Antiretroviral Therapy-Naive Patients with Isolated Nonnucleoside Reverse Transcriptase Inhibitor-Associated Transmitted Drug Resistance. J Acquir Immune Defic Syndr. 2016; 72:171-6.
de Mendoza C, Anta L, García F, et al. HIV-1 genotypic drug resistance interpretation rules - 2009 Spanish guidelines. AIDS Rev. 2009; 11:39-51.
HIVdb Program. Genotypic Resistance Interpretation Algorithm. Sierra version 2.2.5 (last updated 2018-04-10). HIVdb version 8.4 (last updated 2017-06-16) https://hivdb.stanford.edu/hivdb/by-mutations/report/.
Wensing AM, Calvez V, Günthard HF, et al. Update of the Drug Resistance Mutations in HIV-1. Top Antivir Med. 2017; 24:132-133.
Genotype interpretation: non-nucleoside reverse transcriptase inhibitors ANRS - ac 11: resistance group, September 2017- Version n°27. http://www.hivfrenchresistance.org/2017/tab1.html
Deshpande A, Jauvin V, Magnin N, et al. Resistance mutations in subtype C HIV type 1 isolates from Indian patients of Mumbai receiving NRTIs plus NNRTIs and experiencing a treatment failure: resistance to AR. AIDS Res Hum Retroviruses 2007; 23:335-40
8. INTERPRETACIÓN DE RESISTENCIA A IP
Autores: Miguel García Deltoro, Carmen Ricart Olmos
La proteasa es una enzima codificada por una secuencia de 99 aminoácidos ubicada en el gen pol que ejerce su acción en las etapas tardías de la replicación viral. Se encarga del procesamiento o escisión de las poliproteínas gag y gag-pol, paso necesario para convertir las nuevas partículas virales nacientes inmaduras no infecciosas, en partículas virales maduras capaces de infectar a otras células.12
Los IPs compiten con el sustrato natural de la proteasa del VIH en su unión al centro activo, al tener una estructura química similar a los puntos de la cadena peptídica donde el enzima produce los cortes.3 Pero, además en la actualidad, se conoce que el efecto de bloqueo de los IP va más allá, y se da en cada paso relevante del ciclo de vida del VIH incluidas la entrada, la transcripción inversa, la integración y en los procesos post integración al favorecer la formación de moléculas de integrasa, transcriptasa inversa y proteasa disfuncionantes.4
Debemos recordar dos conceptos básicos para poder interpretar la dinámica de resistencias de los farmacos antirretrovirales, y concretamente de los IP:
- Barrera genética: es el número de mutaciones de resistencia necesarias para que el virus desarrolle resistencia fenotípica a ese fármaco
- Robustez: es la dificultad para que aparezcan mutaciones de resistencia cuando se produce el fracaso virológico en presencia del fármaco.
Los IP, cuando están potenciados, son prácticamente los únicos fármacos que conjugan ambas virtudes: tienen una alta barrera genética, y así, para que un virus adquiera resistencia significativa se requiere la acumulación de varias mutaciones de resistencia; y una alta robustez, puesto que es excepcional la selección de resistencias en el fracaso virológico, especialmente en pacientes en su primera línea de tratamiento.5 Además, otra característica de los IP potenciados es que protegen frente a la selección de mutaciones de resistencia a los ITIAN acompañantes, observando sólo en casos aislados la mutación M184I/V. En el manejo de un fracaso a una pauta de inicio con IP potenciados con 2 ITIAN sin mutaciones de resistencia, se puede mantener el mismo TAR. En un estudio combinado realizado con los fracasos de 3 ensayos aleatorizados realizados por los ACTG se concluye que es igual de eficaz que cambiar a una pauta de rescate con fármacos antirretrovirales de otras familias, entre los factores asociados a la posterior supresión virológica destaca sobre todo la mejora de la adherencia.6
Se puede por lo tanto concluir que los IPs potenciados son los fármacos con mayor permisividad a los fallos de adherencia. Farmacológicamente, la alta barrera genética y de resistencia se sustentan, primero en un coeficiente inhibitorio muy elevado frente a la IC50 del virus wild-type,7 segundo en una semivida disociativa muy prolongada8 y, por último, quizás en el comentado efecto multi-step.4
Nada menos que más de 70 mutaciones localizadas en 28 codones del gen de la proteasa, se han asociado en mayor o menor grado con resistencia a los IP.910
Las mutaciones de resistencia a IP pueden ser: 9
- Mutaciones Primarias, Principales o Mayores: relacionadas directamente con la unión de la proteasa al IP, y que contribuyen en mayor medida a la resistencia.
- Mutaciones Secundarias, Menores o Compensatorias: no están directamente relacionadas con la unión al IP, mejoran la capacidad replicativa de los virus mutantes resistentes teniendo un menor efecto, de tal forma que sólo disminuyen la sensibilidad si se asocian a resistencias primarias.
- Polimorfismos Naturales: cerca del 50% de los pacientes que nunca han recibido IP tiene algunas mutaciones secundarias, que no contribuyen a disminuir la sensibilidad en ausencia de otras mutaciones.
Las instituciones europeas y americanas más conocidas como ANRS11 Rega,12 Geno2pheno,13 IAS-USA10, y Standford9 proponen diferentes algoritmos de interpretación para determinar el grado de resistencia a los ARV y particularmente de los IP. Nosotros lo hemos hecho desde la actualización de la Red de Investigación en SIDA (RIS) de enero del año 202014 proponiendo una guía práctica y sencilla, para utilizar en la clínica diaria sin tener que poner los datos en ninguna plataforma informática y evitando puntuaciones complejas para cada una de las mutaciones.9101112 La adaptación, la hemos realizado fundamentalmente de la ANRS,11 pero teniendo en cuenta también mutaciones que, a pesar de no estar en algunos scores de los fármacos,151617 sí que tienen varias referencias que muestran su relevancia en la resistencia18192021y, por ejemplo, así figuran claramente en la base de Stanford y en otras plataformas colaborativas.910111213141516171819202122
Dado que en la actualidad únicamente se utilizan tres IPs: LPV, ATV y DRV, en la tabla adjunta resumen de interpretación de resistencias a los IPs Tabla 1, se definen tan solo las resistencias a esta triada. Sin embargo, entendiendo que hay pacientes que han fracasado a los IPs de primera generación, para ayudar a identificar las posibles mutaciones de resistencias que algunos pacientes pudieran haber seleccionado, sobre todo si en su día no se pudieron determinar, comentaremos muy brevemente las vías de escape o mutaciones más frecuentes y sus características: indinavir: M46L, V82A, I54V; ritonavir: M46L, V82A; saquinavir: G48V, L90M; nelfinavir: D30N, L90M; amprenavir /fosamprenavir: V32I,I47V,I50L,I84V.
DRV, es el que presenta la mayor barrera a las resistencias, no solo de los IPs sino de todo el arsenal terapéutico antirretroviral actual, además es el único que puede aumentar su dosis en pacientes con resistencias previas. Según datos obtenidos en el estudio EMERALD, los pacientes con fallo virológico previo y datos basales de archivo genotípico que recibieron TAF/FTC/DRV/c, las mutaciones de resistencia archivadas a DRV, FTC y TFV no impidieron la respuesta virológica.23LPV tendría una barrera a las resistencias intermedia, aunque actualmente se utiliza menos y, por último, ATV es el que tendría una barrera a las resistencias más baja, especialmente cuando se utiliza no potenciado.
Puede verse claramente Tabla 1, que tanto el ATV como el LPV pueden tener una resistencia fenotípica de alto nivel con tan solo ciertas mutaciones únicas, o bien con el acúmulo de otras varias. DRV necesita al menos dos o bien una si es de las de mayor peso (I50V, I54L/M, L76V o I84V) para su formulación de 800 mg una vez al día (qd) y, al menos cuatro para la de 600 mg dos veces al día (bid) o bien tres si son de las referidas de mayor impacto fenotípico17. Por otra parte, la presencia de mutaciones que producen hipersusceptibilidad242526 junto con mutaciones de resistencia, las neutralizarían en parte confiriendo una resistencia tan solo intermedia.
Por último, hay que destacar que la mutación V32I es una sustitución clave que rara vez ocurre en los intentos de selección in vitro, pero que una vez lo hace, predispone a VIH-1 a desarrollar resistencia a DRV de alto nivel.27 V32I generalmente ocurre junto a mutaciones en el codón 47. En combinación con I47V, provoca una resistencia intermedia tanto para LPV como para DRV y sienta las bases para niveles más altos de resistencia. Por último, junto a I47A provoca resistencia LPV de alto nivel y resistencia a DRV intermedia.9 Habrá que revisar los escasos aislados in vivo de alto impacto fenotípico a DRV para ver si la contienen.
Tablas:

LPV: Lopinavir. Rtv: Ritonavir. ATV: Atazanavir. DRV: Darunavir. Cob: Cobicistat. Bid: dos veces al día. Qd: Una vez al día. *LPV y ATV pueden tener resistencia de alto nivel con mutaciones unitarias o bien con el acúmulo de varias; para el ATV no potenciado con cualquiera de las mutaciones aisladas que aparecen en su recuadro ya se puede considerar resistencia de alto nivel. **En presencia de dos o más mutaciones a DRV o bien de una de las señaladas en negrita (I50V,I54L/M,L76V o I84V) se debe elegir la dosificación de 600 mg bid; asimismo con 3 ó más de las señaladas en negrita ya se considerará resistencia de alto nivel para la dosificación de 600 mg bid16. ***La presencia de mutaciones de hipersusceptibilidad junto a las de resistencia, tendría una interpretación de resistencia intermedia.
Bibliografía:
Flexner Ch. HIV-Protease inhibitors. N Engl J Med. 1998; 338:1281-92
Adamson CS. Protease-Mediated Maturation of HIV: Inhibitors of protease and the maturation process. Mol Biol Int 2012; 2012:604261.
Davis DA, Soule EE, Davidoff KS, et al. Activity of human immunodeficiency virus type 1 protease inhibitors against the initial autocleavage in Gag-Pol polyprotein processing. Anitimicrob Agents Chemother 2012; 56:3620-8
Rabi SA, Laird GM, Durand CM, et al. Multi-step inhibition explains HIV-1 protease inhibitor pharmacodynamics and resistance.J Clin Invest 2013; 123:3848-3860.
Blanco Arévalo JL. Darunavir as initial therapy for human immunodeficiency virus infection and as a strategy for switching regimen not motivated by virological failure. Enferm Infecc Microbiol Clin. 2016; 34 Suppl 1:3-10.
Zheng Y, Hughes MD, Lockman S, et al. Antiretroviral therapy and efficacy after virologic failure on first-line boosted protease inhibitor regimens. Clin Infect Dis 2014; 59:888-96.
Tang MW, Shafer RW. HIV-1 antiretroviral resistance: scientific principles and clinical applications. 2012; 72:1-25
Dierynck I, De Wit M, Gustin E, et al. Binding kinetics of darunavir to human immunodeficiency virus type 1 protease explain the potent antiviral activity and high genetic barrier. J Virol 2007; 81:13845-51.
Standford HIV Drug Resistance Database. Major HIV-1 Drug resistance Mutations. Version 9.0 Updated Mar 1, 2021. Disponible en: https://hivdb.stanford.edu/dr-summary/resistance-notes/PI.
Wensing AM, Calvez V, Ceccherini-Silberstein F, et al. 2019 update of the drug resistance mutations in HIV-1. Top Antivir Med. 2019;27(3):111-121.
ANRS AC11 Resistance Group. HIV-1 genotypic drug resistance interpretations algorithm’s. October 2021, version Nº32. Disponible en: https://hivfrenchresistance.org/wp-content/uploads/2021/10/Algo2021_v32.pdf
Camacho R, Van Laethem K, Geretti AM, et al. Algorith for the use of genotypic HIV-1 resistance data. Rega v.9.1.0 October2013. Disponible en: https://rega.kuleuven.be/cev/avd/files/software/rega_algorithm/Rega_HIV1_Rules_v9.1.0.pdf
Genotypic Resistance-Algorithm Deutschland. August 2015. Disponible en: http://www.hiv-grade.de/cms/grade/hiv2eu
Guía de resistencias a los antirretrovirales: actualización 2020. Red de investigación en SIDA (RIS). RD16/0025. Disponible en: https://www.redris.es/blog/-/blogs/guia-de-resistencias-a-los-antirretrovirales-de-la-ris-actualizacion-2020
Marcelin AG, Cohen-Codar I, King MS, et al. Virological and pharmacological parameters predicting the response to lopinavir-ritonavir in heavily protease inhibitor-experienced patients. Antimicrob Agents Chemother. 2005; 49:1720-6.
Vora S, Marcelin AG, Günthard HF, et al. Clinical validation of atazanavir/ritonavir genotypic resistance score in PI-experienced patients. AIDS 2006; 20:35-40.
De Meyer S, Dierynck I, Lathouwers E, et al. Phenotypic and genotypic determinants of resistance to darunavir: analysis of data from treatment-experienced patients in POWER 1, 2, 3 and DUET-1 and 2. XVII International HIV drug resistance workshop: basic principles and clinical implications, 10-14 June 2008, Sitges, Spain, abstract 31.
De Mendoza C, Valer L, Bacheler L, et al. Prevalence of the HIV-1 protease mutation I47A in clinical practice and association with lopinavir resistance. AIDS 2006; 20: 1071-4.
Nijhuis N, Wensing AMJ, Bierman W, et al. A novel genetic pathway involving L76V and M46I leading to lopinavir/r resistance. XVI International HIV drug resistance workshop: basic principles and clinical implications, 12-16 June 2007, Barbados, West Indies, abstract 127.
Barber TJ, Harrison L, Asboe D, et al. Frequency and patterns of protease gene resistance mutations in HIV-infected patients treated with lopinavir/ritonavir as their first protease inhibitor. J Antimicrob Chemother 2012; 67:995-1000.
Colonno RJ, Rose R, McLaren C, et al. Identification of I50L as the signature atazanavir (ATV)-resistance mutation in treatment naïve HIV-1 infected patients receiving ATV-containing regimens. JID 2004; 189: 1802-10
Paredes R, Tzou PL, van Zyl G, et al. Collaborative update of a rule-based expert system for HIV-1 genotypic resistance test interpretation. PLoS One 2017 12: e0181357.
Lathouwers E, Weinsteiger S, Baugh B, et al. Week 96 resistance analyses of the once-daily,single-tablet regimen (STR) darunavir/cobicistat/emtricitabine/tenofovir alafenamide (D/C/F/TAF) in adults living with HIV-1 from the phase 3 randomized AMBER and EMERALD trials. J Med Virol. 2021 Jun; 93(6):3985-3990.
Weinheimer S, Discotto L, Friborg J, et al. Atazanavir signature I50L resistance substitution accounts for unique phenotype of increased susceptibility to other protease inhibitors in a variety of human immunodeficiency virus type 1 genetic backbones. Antimicrob Agents Chemother 2005.
Wiesmann F, Vachta J, Ehret R, et al. The L76V mutation in HIV-1 protease is potentially associated with hypersusceptibility to protease inhibitors Atazanavir and Saquinavir: is there a clinical advantage? AIDS Res Ther 2011.
Resch W, Ziermann R, Parkin N, et al. Nelfinavir-resistant, amprenavir-hypersusceptible strains of human immunodeficiency virus type 1 carrying an N88S mutation in protease have reduced infectivity, reduced replication capacity, and reduced fitness and process the Gag polyprotein precursor aberrantly. J Virol 2002. 76:8659-66.
Aoki M, Das D, Hayashi H, et al. Mechanism of Darunavir (DRV)'s High Genetic Barrier to HIV-1 Resistance: A Key V32I Substitution in Protease Rarely Occurs, but Once It Occurs, It Predisposes HIV-1 To Develop DRV Resistance. M Bio. 2018; 9: e02425-17.
9. INTERPRETACIÓN DE RESISTENCIA A LOS INHIBIDORES DE LA INTEGRASA
Federico García y Arkaitz Imaz
Los INIs de primera generación, RAL y EVG, son fármacos con baja barrera para el desarrollo de resistencia mientras que los de segunda generación, DTG y BIC presentan una alta barrera a la resistencia. Cabotegravir es un nuevo INIs con una estructura similar a DTG, pero con menor barrera a la resistencia que éste.
Resistencia a Raltegravir y Elvitegravir
Existen tres rutas documentadas de adquisición de resistencia a RAL: la vía de la mutación Y143R/C, la de N155H, y la de Q148H/K/R1. Generalmente, tienen un impacto importante sobre la capacidad replicativa (CR) del virus, pero si se mantiene la presión de RAL, aparecen nuevas mutaciones que, además de incrementar el nivel de resistencia, son compensatorias de la CR: (i) N155H se acompaña de L74M, E92Q, E97A; (ii) Q148 H/K/R de E138K, G140A/S; (iii) Y143R/C de E157Q, T66A, E92Q. La mutación N155H suele ser la primera y la más frecuente en aparecer; si se mantiene la presión farmacológica esta vía evoluciona hacia la selección de las mutaciones Q148H y G140S, que confieren un alto grado de resistencia fenotípica con una CR similar a la del virus salvaje.
Las mutaciones de resistencia seleccionadas tras el fracaso con EVG incluyen cambios en 6 codones de la integrasa: T66I/A/K, E92Q/G, T97A, S147G, Q148H/R/K y N155H. En general T66I/A se selecciona de forma precoz, E92Q y S147G de forma simultánea o sucesiva y Q148R/K es más tardía, aunque puede aparecer en combinación con las anteriores. Estas mutaciones primarias tienen diferente impacto en la resistencia a EVG in vitro (Q148R > E92Q > N155H > T66I > S147G > T97A) y reducen tanto la susceptibilidad a EVG como la CR. Sin embargo, el mantenimiento de la presión de EVG favorece la selección de otras mutaciones compensatorias que confieren mayor grado de resistencia e incrementan la CR, como E138A/K, G140C/S o S147G.
Las mutaciones en el codón 143 (Y143R/C) no tienen prácticamente impacto en la resistencia fenotípica a EVG. Del mismo modo, las mutaciones T66I/A E92G apenas impactan, por si solas, en la resistencia a RAL. Sin embargo, dada la similitud en los perfiles de resistencia de ambos fármacos, no se recomienda el uso EVG tras fracaso a RAL y viceversa345.
Es extremadamente importante detectar precozmente los fracasos con RAL y EVG para retirarlos lo antes posible y evitar la acumulación de mutaciones (lo que podría permitir en algunos casos conservar la actividad de INI de 2ª generación).
Resistencia a Dolutegravir
Las mutaciones seleccionadas por cepas VIH-1 “salvajes” expuestas a DTG in vitro, fundamentalmente R263K67 –G118R en subtipos C- y menos frecuentemente S153F, S153Y, L101/IS153F, T124A/S153Y, y L101IT/124A/S153F, confieren bajo nivel resistencia a DTG, y reducen de forma marcada la Capacidad Replicativa (CR) e infectiva del virus8.
En ensayos clínicos en pacientes naïve, la selección de mutaciones en aquellos pacientes que presentaron fracaso virológico91011 fue irrelevante, y en la práctica clínica habitual, la comunicación de selección de resistencias tras un fracaso a una primera línea de TAR con DTG ha sido excepcional12131415
En el ensayo clínico SAILING, en pacientes con fracasos previos a otras pautas de TAR y resistencia a al menos 2 clases de ARV pero naïve a INIs, se describió la selección de R263K -junto con la mutación polimórfica C151I- en un paciente, y N155H en otros dos, tras el fracaso con un régimen que incluía DTG16.
DTG mantiene su actividad in vitro frente a variantes virales resistentes a RAL y EVG con una única mutación, así como frente a la asociación de E92Q, Y143C/R, o N155H con otras mutaciones secundarias. Solo la combinación de una mutación en la posición 148 junto con otras mutaciones secundarias confiere resistencia a DTG17.
En los ensayos clínicos VIKING, en pacientes con fracaso a INIs de primera generación, se observó que una mutación en el codón 148 junto con al menos otras 2 mutaciones –que incluyen fundamentalmente cambios en la posición 140- es el principal predictor de respuesta a DTG, asociándose de forma significativa con menor respuesta tras 7 días monoterapia con DTG y menor probabilidad de supresión viral tras 24 semanas de tratamiento con DTG más una pauta con al menos otro fármaco activo1819.
Resistencia a Bictegravir
El perfil de resistencia de BIC muestra bastante similitud con DTG desde el punto de vista de las posiciones implicadas20. Las mutaciones seleccionadas in vitro son R263K±M50I o S153F/Y, y también tienen un bajo impacto en la actividad de BIC. De forma similar a DTG, BIC mantiene actividad antiviral in vitro frente a variantes resistentes a RAL y EVG con mutaciones únicas. Ante dobles mutantes (E138K/Q148K, G140S/Q148R, E92Q/N155H y Q148R/N155H) la actividad de BIC también está reducida, si bien el impacto de algunas de estas combinaciones sobre BIC parece ser in vitro algo menor que sobre DTG2021, lo que podría explicarse por un mayor coeficiente de disociación de la integrasa de BIC frente a DTG22. La combinación E92Q/G140E confiere alto nivel de resistencia a todos los INIs incluido BIC aunque su impacto sobre la CR hace poco probable la viabilidad viral in vivo21.
Resistencia a Cabotegravir
A pesar de ser un análogo estructural de DTG, CAB muestra menor barrera a las resistencias que DTG y BIC, pero mayor que RAL y EVG23. En este estudio, las mutaciones seleccionadas in vitro por CAB en distintos aislados fueron H51Y, L74M, E138K, G140S, S147G, Q148K/R, S153A/Y/F, G163R y R263K. El análisis de los diferentes ensayos clínicos en los que se ha utilizado CAB en combinación con RPV no ha demostrado que ninguna mutación específica tenga un papel importante en el fracaso y la generación de resistencias.
Ver Tabla 1
Tablas:
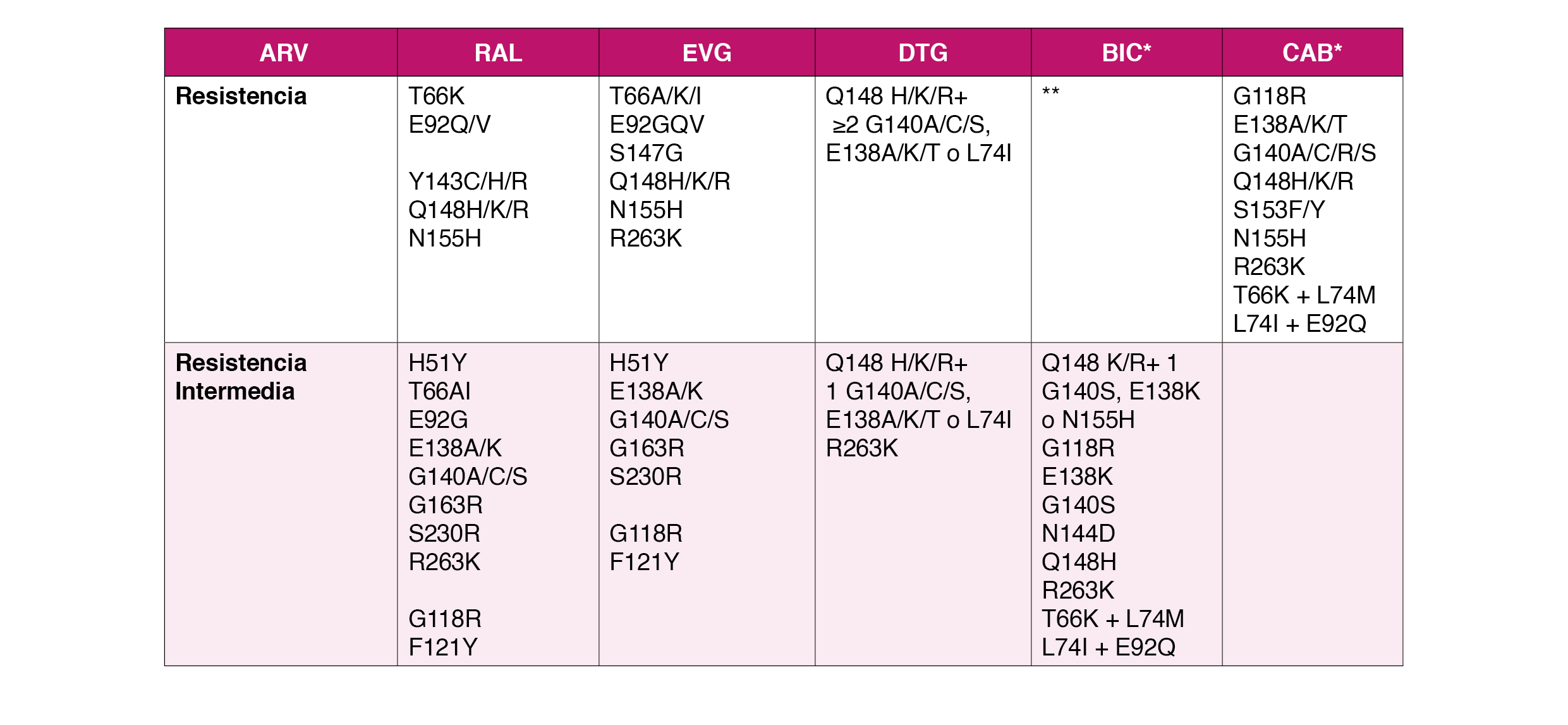
Adaptada de Guía de Resistencias a los Antiretrovirales (RIS)
* Adaptado de https://hivfrenchresistance.org/hiv-french-resitance-integrase-strand-transfer-inhibitors/ y
** Aunque no existen estudios clínicos que hayan evaluado la actividad de BIC en pacientes con fracaso previo y resistencia a otros INI puede considerarse que las combinaciones de mutaciones que confieren resistencia de alto nivel a DTG afectaran de igual manera la actividad de BIC
Los aislados VIH-2 son sensibles a RAL, EVG y DTG, las principales mutaciones asociadas con resistencias a INI en VIH-2 son: G118R, Y143C, Q148K/R, N155H, G140S+Q148R.
Bibliografía:
Thierry E, Deprez E, Delelis O. Different Pathways Leading to Integrase Inhibitors Resistance. Front Microbiol 2017; 7:2165.
Doyle T, Dunn DT, Ceccherini-Silberstein F, et al, Coronet Study. Integrase inhibitor (INI) genotypic resistance in treatment-naive and raltegravir-experienced patients infected with diverse HIV-1 clades. J Antimicrob Chemother 2015; 70:3080-6.
Abram ME, Hluhanich RM, Goodman DD, et al. Impact of primary elvitegravir resistance-associated mutations in HIV-1 integrase on drug susceptibility and viral replication fitness. Antimicrob Agents Chemother 2013; 57:2654-63.
Goethals O, Clayton R, Van Ginderen M, et al. Resistance mutations in human immunodeficiencyvirus type 1 integrase selected with elvitegravir confer reduced susceptibility to a wide range of integrase inhibitors. J Virol 2008; 82:10366-74.
Geretti AM, Armenia D, Ceccherini-Silberstein F. Curr Opin Infect Dis 2012; 25:67786.
Quashie PK, Mesplède T, Han YS, et al. Characterization of the R263K mutation in HIV-1 integrase that confers low-level resistance to the second-generation integrase strand transfer inhibitor dolutegravir. J Virol 2012; 86:2696-705.
Mesplède T, Quashie PK, Osman N, et al. Viral fitness cost prevents HIV-1 from evading dolutegravir drug pressure. Retrovirology 2013; 10:22.
Kobayashi M, Yoshinaga T, Seki T, et al. In Vitro antiretroviral properties of S/GSK1349572, a next-generation HIV integrase inhibitor. Antimicrob Agents Chemother. 2011; 55:813-21.
Raffi F, Jaeger H, Quiros-Roldan E, et al; extended SPRING-2 Study Group. Once-daily dolutegravir versus twice-daily raltegravir in antiretroviral-naïve adults with HIV-1 infection (SPRING-2 study): 96 week results from a randomised, double-blind, non-inferiority trial. Lancet Infect Dis 2013; 13:927-35.
Walmsley S, Baumgarten A, Berenguer J, et al. Brief Report: Dolutegravir Plus Abacavir/Lamivudine for the Treatment of HIV-1 Infection in Antiretroviral Therapy-Naive Patients: Week 96 and Week 144 Results From the SINGLE Randomized Clinical Trial. J Acquir Immune Defic Syndr 2015; 70:515-9.
Molina JM, Clotet B, van Lunzen J, et al; FLAMINGO study team. Once-daily dolutegravir versus darunavir plus ritonavir for treatment-naive adults with HIV-1 infection (FLAMINGO): 96 week results from a randomised, open-label, phase 3b study. Lancet HIV 2015; 2:e127-36.
Lepik KJ, Harrigan PR, Yip B, et al. Emergent drug resistance with integrase strand transfer inhibitor-based regimens. AIDS. 2017; 31:1425-34.
Fulcher J, Du Y, Sun R, et al. Emergence of integrase resistance mutations during initial therapy with TDF/FTC/DTG. Conference on Retroviruses and Opportunistic Infections. February 13–16, 2017. Seattle, Washington. #LB 500.
Pena M, Chueca N, Hernández-Febles M, et al. Use of Next Generation Sequencing to study a first line failure to a Dolutegravir containing regimen. 16th European Meeting on HIV & Hepatitis 2018; Rome, May 2018. #31.
Lübke N, Jensen B, Hüttig F, et al. Failure of Dolutegravir-containing First-line regimen. 16th European Meeting on HIV & Hepatitis 2018; Rome, May 2018. #30.
Cahn P, Pozniak AL, Mingrone H, et al. Dolutegravir versus raltegravir in antiretroviral-experienced, integraseinhibitor-naive adults with HIV: week 48 results from the randomised, doubleblind,non-inferiority SAILING study. Lancet 2013; 382:700-8.
Seki T, Suyama-Kagitani A, Kawauchi-Miki S, et al. Effects of raltegravir or elvitegravir resistance signature mutations on the barrier to dolutegravir resistance in vitro. Antimicrob Agents Chemother 2015; 59:2596-606.
Eron JJ, Clotet B, Durant J, et al. Safety and efficacy of dolutegravir in treatment-experienced subjects with raltegravir-resistant HIV type 1 infection: 24-week results of the VIKING Study. J Infect Dis 2013; 207:740-8.
Castagna A, Maggiolo F, Penco G, et al; VIKING-3 Study Group. Dolutegravir in antiretroviral-experienced patients with raltegravir and/or elvitegravir-resistant HIV-1: 24-week results of the phase III VIKING-3 Study. J Infect Dis 2014; 210:354-62.
Tsiang M, Jones GS, Goldsmith J, et al. Antiviral Activity of Bictegravir (GS-9883), a Novel Potent HIV-1 Integrase Strand Transfer Inhibitor with an Improved Resistance Profile. Antimicrob Agents Chemother 2016; 60:7086-97.
Andreatta K, Chang S, Martin R, et al. Integrase Inhibitor Resistance Selections Initiated with Drug Resistant HIV-1. Conference on Retroviruses and Opportunistic Infections, Boston, MA, March 4-7, 2018. Abstract 546.
White K, Niedziela-Majka A, Novikov N, et al. Bictegravir Dissociation Half-life from HIV-1 G140S/Q148H Integrase-DNA Complexes. Conference on Retroviruses and Opportunistic Infections, February 13-16, 2017, Seattle, WA. #497.
Oliveira M, Ibanescu RI, Anstett K, et al. Selective resistance profiles emerging in patient-derived clinical isolates with cabotegravir, bictegravir, dolutegravir, and elvitegravir. Retrovirology. 2018;15(1):56. Published 2018 Aug 17. doi:10.1186/s12977-018-0440-3
10. RESISTENCIAS A NUEVOS FÁRMACOS EN EL CONTEXTO DE LA MULTIRRESISTENCIA
Autores: Eva Poveda y Hortensia Álvarez
En la actualidad se encuentran en desarrollo nuevos fármacos que presentan mecanismos de acción únicos y que actúan a diferentes niveles del ciclo de replicación del VIH, lo que les permite ser activos frente a aislados resistentes a las familias clásicas de antirretrovirales. Están diseñados por tanto para el tratamiento de personas que viven con VIH multirresistente y con limitadas opciones de tratamiento supresor, en combinación con terapia optimizada de base. En este capítulo, nos centraremos en definir las características y el perfil de resistencia de aquellos que ya están aprobados o que lo estarán a corto plazo Tabla 1.
Ibalizumab
Es un fármaco de acción prolongada (long-acting) aprobado por la FDA y la EMA. Se administra vía intravenosa en una única dosis de 2000 mg seguida de dosis de mantenimiento de 800 mg cada 2 semanas.123
Se trata de un anticuerpo monoclonal que tiene como diana el receptor celular CD4 al que se une en el segundo dominio extracelular, impidiendo que se produzcan los cambios conformacionales inducidos tras la unión de la glicoproteína gp120 de la envuelta del VIH por un impedimento estérico y por tanto, la entrada del virus en la célula.
Presenta actividad frente a variantes R5 y X4-trópicas, por tanto, su actividad es independiente del tropismo viral y no presenta resistencia cruzada a otras familias de antirretrovirales como los ITIAN, ITINAN, IP, INI, o a los inhibidores de la entrada Enfuvirtida o MVC. Además, presenta actividad frente a un amplio espectro de variantes genéticas del VIH-1 grupo M (e.j. subtipos A, B, C, D, E, O), y frente al VIH-2 (datos in vitro).4
En cuanto al perfil de resistencias primarias, los datos disponibles de ensayos fenotípicos realizados hasta el momento en más de 160 aislados virales, han demostrado una alta sensibilidad al fármaco.5
El principal mecanismo de resistencia a IBA identificado hasta el momento es la pérdida de sitios de glicosilación en la envuelta viral, en concreto, en la región variable V5 de gp120, lo que se correlaciona con pérdida de susceptibilidad al fármaco.6
Fostemsavir
Es otro fármaco inhibidor de la entrada del virus en la célula, también aprobado por la FDA7 y la EMA.8Se administra vía oral, en dosis de un comprimido de 600 mg dos veces al día.9
Es un profármaco metabolizado a su forma activa Temsavir que se une directamente a la glicoproteína de la envuelta del VIH gp120, impidiendo así la unión inicial del virus y por tanto su entrada en la célula.
La actividad de FOS es también independiente del tropismo viral y no presenta resistencia cruzada a otras familias de antirretrovirales. Sin embargo, FOS no es activo frente a las formas recombinantes CRF01_AE y a aislados del grupo O, ya que presentan polimorfismos que los hacen naturalmente resistentes al fármaco (S375H y M475I para CRF01_AE).10 FOS tampoco es activo frente al VIH-2.
En el caso de FOS, se han identificado polimorfismos basales en gp120 que podrían comprometer la respuesta al fármaco (S375H/I/M/N/T, 33%; M426L, 12%; M434I, 6%; M475I, 1%). Sin embargo, la respuesta virológica no se pudo predecir totalmente en función de la presencia de estos polimorfismos o el subtipo genético.11 Por lo tanto, son necesarios más datos para conocer mejor el impacto que estos polimorfismos podrían tener en la respuesta virológica.
En el escenario de fracaso a tratamiento con FOS, se han identificado sustituciones genotípicas emergentes en las mismas 4 posiciones en el sitio de unión de gp120 al receptor CD4: S375N, 16%; M426L, 24%; M434M/I, 7%; M475M/I, 6%.9
Otros inhibidores de la entrada como MVC e IBA, que comparten interacciones con gp120 como mecanismo de acción, mantienen la actividad frente a aislados que contienen cambios en las principales posiciones de interés de gp120 (S375, M426 y M475), sin evidenciarse resistencia cruzada.12
Lenacapavir
Es un fármaco long-acting de administración subcutánea cada 6 meses.
Se trata de un inhibidor de la cápside viral y que actúa a diferentes niveles del ciclo de replicación inhibiendo tanto el proceso de desencapsidación como el de encapsidación durante la formación de nuevos viriones, ya que se une entre los monómeros adyacentes que forman la cápside viral.13
La actividad de LEN es independiente del tropismo viral y no presenta resistencia cruzada a otras familias de fármacos. Es activo frente a la mayoría de subtipos de VIH-1 y frente al VIH-2.13
Estudios in vitro han identificado principalmente 5 mutaciones en la cápside, asociadas con pérdida de susceptibilidad aLEN: L56I, M66I, Q67H, K70N, N74D.14
Hay que destacar que no se han observado mutaciones asociadas con resistencias al fármaco en secuencias genéticas procedentes de aislados virales de pacientes tanto naive, como pre-tratados y expuestos o no a IP.15 Sin embargo, algunas de estas mutaciones fueron seleccionadas en pacientes pretratados y con VIH multirresistente, que fracasaron al tratamiento con LEN en el ensayo clínico CAPELLA (M66I, Q67H, K70N/R/S, N74D)16 y también en pacientes naïve que fracasaron a tratamiento con LEN en el ensayo clínico CALIBRATE (Q67H, K70R), con posible mala adherencia subyacente.17
Hasta el momento, la experiencia clínica con estos fármacos es escasa, especialmente si tenemos en cuenta que el número de pacientes incluidos en los ensayos clínicos es reducido, además de presentar características muy heterogéneas en cuanto a la exposición previa a fármacos y la actividad de los fármacos acompañantes. Por lo tanto, necesitamos más experiencia para mejorar nuestro conocimiento sobre sus características, desde el punto de las resistencias.
Tablas:
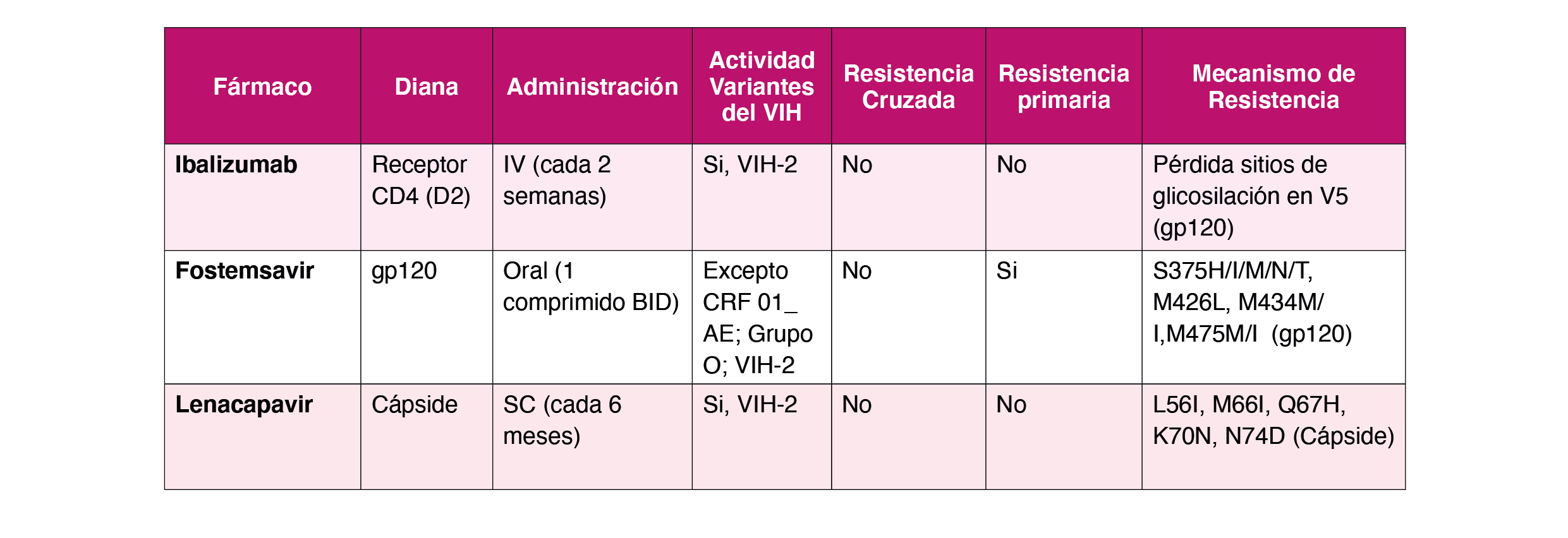
Bibliografía:
Available from https://www.accessdata.fda.gov/drugsatfda_docs/label/ 2018/761065lbl.pdf. Accessed on April 5, 2022.
Available from https://www.ema.europa.eu/en/documents/product-information/trogarzo-epar-product-information_en.pdf. Accessed on April 5, 2022.
Gathe JC., Hardwicke RL., García F., et al. Efficacy, Pharmacokinetics, and Safety Over 48 Weeks With Ibalizumab-Based Therapy in Treatment-Experienced Adults Infected With HIV-1: A Phase 2a Study J Acquir Immune Defic Syndr. 2021; 1;86(4):482-489.
Le Hingrat Q., Collin G., Bachelard A., et al. Ibalizumab shows in vitro activity against group A and group B HIV-2 clinical isolates. AIDS 2022 Mar 8. doi: 10.1097/QAD.0000000000003218.
Jullien H., Leonard M., Cash RB., Mesquita P., Weinheimer S., Marsolais C. Assessment of genotypic patterns associated with HIV-1 sensitivity to Ibalizumab. 18th European Meeting on HIV & Hepatitis. Treatment Strategies &Antiviral Drug Resistance, October 28-30, 2020. Virtual meeting. Abstract 27.
Song R., Oren DA., Franco D., et al. Strategic addition of an N-linked glycan to a monoclonal antibody improves its HIV-1-neutralizing activity. Nat Biotechnol. 2013;31(11):1047-52.
Available from https://www.accessdata.fda.gov/drugsatfda_docs/label/ 2020/212950s000lbl.pdf. Accessed on April 5, 2022.
Available from https://www.ema.europa.eu/en/documents/product-information/rukobia-epar-product-information_en.pdf. Accessed on April 5, 2022.
Lataillade M., Lalezari JP., Kozal M., et al. Safety and efficacy of the HIV-1 attachment inhibitor prodrug fostemsavir in heavily treatment-experienced individuals: week 96 results of the phase 3 BRIGHTE study. Lancet HIV. 2020;7(11):e740-e751.
Zhou N., Nowicka-Sans B., McAuliffe B., et al. Genotypic correlates of susceptibility to HIV-1 attachment inhibitor BMS-626529, the active agent of the prodrug BMS-663068 J Antimicrob Chemother 2014;69(3):573-81.
Gartland M., Ackerman P., Mannino F., et al. Clinical significance of gp120 polymorphisms, TMR IC50FC, and HIV-1 subtype in BRIGHTE. Glasgow, HIV Drug Therapy, October 5-8, 2020.Abstract P019.
Rose B., Gartland M., Stewart E., et al. Reduced susceptibility to temsavir is not linked to IBA or MVC resistance. Conference on Retroviruses and Opportunistic Infections, March 6-10, 2021. Abstract 422.
Yant SR., Mulato A., Hansen D., et al. A highly potent long-acting small-molecule HIV-1 capsid inhibitor with efficacy in a humanized mouse model. Nat Med. 2019;25(9):1377-1384.
Link JO., Rhee MS., Tse WC., et al. Clinical targeting of HIV capsid protein with a long-acting small molecule. 2020;584(7822):614-618.
Marcelin A-G., Charpentier C., Jary A., et al. Frequency of capsid substitutions associated with GS-6207 in vitro resistance in HIV-1 from antiretroviral-naive and -experienced patients. J Antimicrob Chemother. 2020;75(6):1588-1
Molina JM., Segal-Maurer S., Stellbrink H-J., et al. Efficacy and safety of long-acting subcutaneous lenacapavir in phase 2/3 in heavily treatment-experienced people with HIV: week 26 results (Capella study). International AIDS Society, July 18-21, 2021.
Gupta SK., Sims J., Brinson C., et al. Lenacapavir as part of a combination regimen in treatment-naïve people with HIV: week 54 results (Calibrate study). Conference on Retroviruses and Opportunistic Infections, February 12-16, 2022. Oral abstract 138.
11. RESISTENCIAS EN VIH-2 Y SUBTIPOS NO-B
Africa Holguín y Ana López Lirola
El VIH presenta alta variabilidad genética, debida a su alta tasa de replicación, de mutagénesis y de recombinación – a la que también se añade el papel del mecanismo de resistencia innata al VIH-1, APOBEC 3G -, describiéndose numerosas variantes. Se clasifica en dos tipos: VIH-1 y VIH-2. Dentro del VIH - 1 se diferencian por homología genética cuatro grupos: M (main), O (outlier), N (no - M, no - O) y P. A su vez, el grupo M se ha subdividido en 9 subtipos denominados por letras (A, B, C, D, F, G, H, J, K)1 y múltiples recombinantes entre ellos. Existen, al menos 96 CRF y múltiples URF) (https://www.hiv.lanl.gov/content/sequence/HIV/CRFs/CRFs.html)
El subtipo B, variante predominante en Europa y EE.UU., es la más estudiada a nivel virológico, clínico y terapéutico. Sin embargo, los subtipos del VIH-1 distintos al subtipo B y recombinantes (variantes no-B) causan cerca del 90% de los 37 millones de infecciones por VIH.
El VIH-2 es menos prevalente y transmisible que el VIH-12. Infecta a 2 millones de personas, sobre todo en África occidental y zonas con vínculos socioeconómicos afines (Portugal, Francia, España). Su menor virulencia condiciona que más de la mitad de los casos presenten viremias indetectables al diagnóstico3, pero incluso en viremias bajas se han identificado mutaciones de resistencias (MR)4. El VIH-2 no se puede cuantificar con kits de carga viral para VIH-1. Actualmente existe poca evidencia de calidad y falta de comercialización de kits para la monitorización y el genotipado del VIH-2 imprescindible para el manejo de pacientes donde se ha demostrado la presencia de MR primarias transmitidas, la inmunosupresión avanzada y el fracaso virológico tras el tratamiento antirretroviral. Sin embargo, existen algoritmos para identificar las MR en VIH-2 (http//www.hiv-grade.de/HIV2EU)5 y para la interpretación genotípica de resistencias (http://www.hivfrenchresistance.org/2017/Algo2017-HIV2.pdf).
Debido a la alta variabilidad genética, cada tipo, grupo, subtipo o recombinante del VIH presenta polimorfismos naturales a lo largo de su genoma fijados durante la evolución viral, que son específicos de una o varias variantes. Algunos se localizan en residuos asociados a la resistencia a fármacos antirretrovirales (ARV)6789. Cuando estos polimorfismos naturales coinciden con MR primarias aparece resistencia natural a ciertos ARV, limitando el arsenal terapéutico para las variantes que lo presentan.
Así, el VIH-2 presenta resistencia natural a ITINAN por la presencia natural de Y181I/V, Y188L y G190A en su retrotranscriptasa10, a inhibidores de la fusión por los cambios N42Q y N43Q en gp41 o a IPs (amprenavir, tipranavir y ATV) por las sustituciones I32V, V47I, M76L, e I82V en la proteasa viral11. El VIH-2 también muestra menor barrera genética para ITIAN, siendo las TAMs más raras y desarrollando rápidamente la K65R, Q151M, M184V en presencia de ITIAN41213.
Aunque se ha demostrado actividad antiviral con cenicriviroc (antagonista dual de cepas CCR5 y CCR2) en aislados del VIH-2 con tropismo CCR514, no se recomienda su uso dada la afinidad por otros correctores del virus, como CCR1, CCR3, CCR6, GPR15 (BOB), CCR8 y CXCR6. Los INI han demostrado eficacia para VIH-2 y presentan un perfil de resistencias similar al VIH-115.
Respecto al VIH-1, el grupo O del VIH-1 presenta resistencia natural a ITINAN por la presencia de Y181C en su TI16. Para el grupo M del VIH-1, la mayoría de polimorfismos naturales asociados a resistencias se localizan en posiciones de resistencia secundaria, lo que podría explicar la misma eficacia terapéutica en pacientes tratados infectados con distintas variantes virales observadas en varios estudios817. Sin embargo, el bajo número de pacientes con ciertas variantes no-B del VIH-1 y el uso de terapias combinadas analizadas en estos trabajos podrían enmascarar una distinta respuesta a ARV individuales, como han observado otros trabajos que comparan eficacia de monoterapias en pacientes con subtipo C18, subtipo F19 o subtipo G y recombinante CRF02_AG20 con respecto al subtipo B.
Es importante destacar que los polimorfismos naturales en posiciones de resistencia en variantes concretas pueden influir en la barrera genética a ciertos ARV21, en las vías de resistencia22, en la mejora de la capacidad replicativa o fitness del virus bajo presión farmacológica23, en la susceptibilidad a ARV específicos924 o alterar la interpretación de la resistencia a ciertos ARV utilizando diferentes algoritmos1625. Asimismo, pueden favorecer la aparición más rápida de mutaciones de resistencia primaria en variantes concretas debido al uso de tripletes de nucleótidos diferenciales para ese residuo relacionado con resistencia en esas variantes virales26. Por ejemplo, la MR a ITINAN V106M ocurre con mayor frecuencia en virus de subtipo C de pacientes tratados con NVP o EFV porque V106M requiere un cambio de un nucleótido en el subtipo C [GTG (V) => ATG (M)], pero un cambio de dos pares de bases en otras variantes [GTA (V) => ATG (M)]26. Un mecanismo similar explicaría la aparición preferente de la MR a ITIAN V75M en los recombinantes CRF01_AE27, de la MR a IPs V82M en virus del subtipo G28, y de la MDR a ITINAN G190S en los subtipos A de la antigua Unión Soviética29. Varias líneas de evidencia también sugieren que K65R es más probable y rápido que se seleccione en los virus de subtipo C que en otros subtipos26.
Ver Tabla 1
Tablas:
Bibliografía:
Peeters M, Jung M, Ayouba A. The origin and molecular epidemiology of HIV. Expert Rev Anti Infect Ther 2013; 11:885-96.
Clavel F, Guyader M, Guétard D, et al. Molecular cloning and polymorphism of the human immune deficiency virus type 2. Nature 1986; 324:691-5.
Popper SJ, Sarr AD, Travers KU, et al. Lower human immunodeficiency virus (HIV) type 2 viral load reflects the difference in pathogenicity of HIV-1 and HIV-2. J Infect Dis 1999; 180: 1116-21.
Gottlieb G,Dia Badiane NM,Hawes SE, et al. Emergence of multiclass drug-resistance in HIV-2 in antiretroviral-treated individuals in Senegal: Implications for HIV-2 tratment in resource-limited West Africa. Clin Infect Dis 2009; 48:476-83.
Charpentier Ch, Camacho R, Ruelle J et al, HIV-2EU: Supporting Standarized HIV-2 Drug Resistence interpretation in Europe. Clin Infect Dis 2013;56:1654-8.
Llácer Delicado T, Torrecilla E and Holguín A. Deep analysis of HIV-1 natural variability across HIV-1 variants at residues associated with integrase inhibitors (INI) resistance in INI-naïve individuals. J Antimicrob Chemother 2016; 71:362-6.
Wainberg MA, Brenner BG. The impact of HIV genetic polymorphisms and subtype differences on the occurrence of resistance to antiretroviral drugs. Mol Biol Int 2012:256982.
Lessells RJ, Katzenstein DK, de Oliveira T. Are subtype differences important in HIV drug resistance? Curr Opin Virol 2012; 2:636-43.
Holguín A, Ramírez de Arellano E, Rivas P y Soriano V. Efficacy of antiretroviral therapy in individuals infected with HIV-1 non-B subtypes. AIDS Rev 2006; 8:98-107.
Witvrouw M, Pannecouque C, Switzer WM, Folks TM, De Clercq E, Heneine W. Susceptibility of HIV-2, SIV and SHIV to various anti–HIV-1 compounds: implications for treatment and postexposure prophylaxis. Antiviral Therapy 2004; 9:57-65.
Raugi DN, Smith RA, Ba S, et al. Complex patterns of protease inhibitor resistence among antiretroviral treatment-experienced HIV-2 patients from Senegal: Implications for second-line therapy. Antimicrob Agent Chemother 2013; 57:2751-60.
Smith RA, Anderson DJ, Pyrak CL, et al. Antiretroviral drug resistance in HIV-2: three amino acid changes are sufficient for classwide nucleoside analogue resistance. J Infect Dis 2009;199:1323-6.
Descamps D, Damond F, Matheron S, et al. High frequency of selection of K65R and Q151M mutations in HIV-2 infected patients receiving nucleoside reverse transcriptase inhibitors containing regimen. J Med Virol 2004; 74:197-201.
Visseaux B, Charpentier C, Collin G, et al. Cenicriviroc, a Novel CCR5 (R5) and CCR2 Antagonist, Shows In Vitro Activity against R5 Tropic HIV-2 Clinical Isolates. PLoS One 2015;10:e0134904.
Treviño A, Cabezas T, Lozano A eta al. Dolutegravir for the treatment of HIV-2. J Clin Virol 2015; 64:12-5
Depatureaux A, Charpentier C, Leoz M, et al. Impact of HIV-1 group O genetic diversity on genotypic resistance interpretation by algorithms designed for HIV-1 group M. J Acquir Immune Defic Syndr 2011; 56:139-45.
Touloumi G, Pantazis N, Chaix ML, et al. Virologic and immunologic response to cART by HIV-1 subtype in the CASCADE collaboration. PLoS One. 2013;8(7):e71174.
Kantor R, Smeaton L, Vardhanabhuti S, et al. Pretreatment HIV drug resistance and hiv-1 subtype c are independently associated with virologic failure: results from the multinational PEARLS (ACTG A5175) Clinical Trial. Clin Infect Dis 2015; 60:1541-9.
Pernas B, Grandal M, Mena A, et al. High prevalence of subtype F in newly diagnosed HIV-1 persons in northwest Spain and evidence for impaired treatment response. AIDS 2014; 28:1837-40.
Sutherland KA, Ghosn J, Gregson J, et al. HIV-1 subtype influences susceptibility and response to monotherapy with the protease inhibitor lopinavir/ritonavir. J Antimicrob Chemother 2015; 70:243-8.
Fofana DB, Charpentier C, Maïga AI, et al. Genetic barrier for attachment inhibitor BMS-626529 resistance in HIV-1 B and non-B subtypes. J Antimicrob Chemother 2015; 70:130-5
Grossman Z, Paxinos EE, Averbuch D, et al. Mutation D30N is not preferentially selected by human immunodeficiency virus type 1 subtype C in the development of resistance to nelfinavir. Antimicrob Agents Chemother 2004; 48:2159-65.
Holguín Á, Sune C, Hamy F, et al. Natural polymorphisms in the protease gene modulate the replicative capacity of non-B HIV-1 variants in the absence of drug pressure. J Clin Virol 2006; 36:264-71.
Poveda E, de Mendoza C, Parkin N, et al. Evidence for different susceptibility to tipranavir and darunavir in patients infected with distinct HIV-1 subtypes. AIDS. 2008; 22:611-6.
Yebra G, de Mulder M, del Romero J, et al. HIV-1 non-B subtypes: High transmitted NNRTI-resistance in Spain and impaired genotypic resistance interpretation due to variability. Antiviral Res 2010; 85:409-17.
Brenner BG, Oliveira M, Doualla-Bell F, et al. HIV-1 subtype C viruses rapidly develop K65R resistance to tenofovir in cell culture. AIDS 2006; 20:F9-13.
Ariyoshi K, Matsuda M, Miura H, et al. Patterns of point mutations associated with antiretroviral drug treatment failure in CRF01_AE (subtype E) infection differ from subtype B infection. J Acquir Immune Defic Syndr 2003; 33:336-42.
Palma AC, Covens K, Snoeck J, et al. HIV-1 protease mutation 82M contributes to phenotypic resistance to protease inhibitors in subtypeG. J Antimicrob Chemother 2012; 67:1075-9.
Kolomeets AN, Varghese V, Lemey P, et al. A uniquely prevalent nonnucleoside reverse transcriptase inhibitor resistance mutation in Russian subtype A HIV-1 viruses. AIDS 2014; 28:F1-8.
12. MANEJO CLÍNICO DEL PACIENTE MULTITRATADO
Rocio Montejano y José Luis Blanco
El tratamiento antirretroviral del paciente multitratado en fracaso virológico, supone uno de los retos más complejos en el manejo del paciente con infección por VIH en la actualidad. Aunque este escenario se presenta actualmente en pocas ocasiones, cuando lo hace exige al clínico un profundo análisis tanto de la historia farmacológica del paciente, como de la interpretación de los patrones de resistencias y de las características de los fármacos existentes. A continuación, analizaremos la epidemiología así como los aspectos principales a considerar en los pacientes en este escenario.
¿Cómo definimos el paciente multitratado?
El paciente HTE ("Highly Treatment Experienced") ha sido clásicamente definido como aquel que presenta resistencia a al menos 1 fármaco antirretroviral de cada una de las 3 clases principales (incluyendo ENF y/o MVC). A partir de la aparición de los INIs, esta definición ha cambiado y mayoritariamente se consideran HTE los individuos con resistencia a al menos 1 fármaco de cada una de las 4 clases principales (incluyendo ENF y/o MVC).1
Sin embargo, esta definición puede no reflejar bien la esencia de intentar caracterizar a estos individuos que sería la de aquellos sujetos en los que no es posible construir un régimen de TAR eficaz por motivos de resistencias, intolerancias u otras consideraciones de seguridad (como comorbilidades o interacciones farmacológicas).
La FDA define a los pacientes multitratados como aquellos con resistencia a múltiples fármacos y clases de fármacos MDR (del inglés "multiple drug resistance") en los que no es posible construir un régimen que suprima el ARN del VIH por debajo de los límites de cuantificación del ensayo.2
Esta situación afecta principalmente a dos grupos de individuos: adultos que se infectaron al principio de la epidemia y niños infectados por transmisión vertical; en ambos grupos los tratamientos eran insuficientes por falta de potencia y/o alta resistencia y se vieron sometidos a tratamientos secuenciales no supresores y con baja barrera a las resistencias que favorecieron la selección de un virus cada vez de mayor complejidad.34
¿Cuál es el objetivo del TAR en el paciente multitratado en fracaso virológico?
El objetivo del TAR para los pacientes HTE&MDR que experimentan un fracaso virológico (FV) tiene que ser inicialmente el mismo que el de cualquier individuo en FV: conseguir la supresión virológica sostenida (es decir, niveles de ARN del VIH por debajo de los límites inferiores de detección).
En el caso de algunos sujetos multitratados y con una gran resistencia a los todos o la mayoría de los fármacos antirretrovirales, puede que no sea posible conseguir una supresión virológica. En este caso, el tratamiento antirretroviral debe diseñarse para: retrasar la progresión clínica, preservar la cifra de linfocitos CD4+ y minimizar la toxicidad.
¿Cómo hay que abordar el diseño del tratamiento de rescate?
A la hora de diseñar un nuevo régimen de TAR para un paciente que haya tenido múltiples fracasos virológicos previos y esté en FV, se deben analizar todas las mutaciones seleccionadas en los diferentes estudios de resistencias genotípicos, así como, si las hubiera, las resistencias fenotípicas identificadas. Del mismo modo se tendrán en cuenta los posibles test de tropismo que se hubieran podido realizar con anterioridad, en caso de que uno de ellos haya mostrado un tropismo no R5 (dual o X4) se considerara que el MVC ya no es activo. A partir de la suma de todas las mutaciones identificadas en todos los test de resistencias disponibles se determinará un score de sensibilidad genotípica acumulado (GSS), siendo la suma aritmética de las puntuaciones individuales de los fármacos específicos el GSS total.5
El GSS ofrece una evaluación más completa de la carga de resistencia, siendo predictor de respuesta virológica.6 Las puntuaciones más altas se han asociado de forma significativa con una mayor probabilidad de supresión virológica, tanto en los ensayos clínicos de los fármacos en investigación como en estudios de cohortes. Este enfoque puede servir de guía para mejorar la selección del TAR para pacientes multitratados.7
No obstante, el genotipo acumulado podría no identificar todas las mutaciones de resistencia seleccionadas por el virus del paciente, ya sea por la no realización de un estudio de resistencias tras el fracaso a un determinado esquema -la falta de presión selectiva de un fármaco en el momento del genotipado evita la identificación de mutaciones seleccionadas que tras la retirada del fármaco pasarían a poblaciones minoritarias que no identifica el test genotípico de secuenciación poblacional, o por la relativa baja sensibilidad de las pruebas genotípicas, siendo capaz de predecir las respuestas al tratamiento con una precisión en torno al 60-65%. 5
¿Qué otras herramientas podemos utilizar: papel de la secuenciación masiva o resistencias en CMSP?
La secuenciación masiva aumenta la sensibilidad de detección de mutaciones de resistencias en poblaciones virales más bajas del 15-20% que identifican los test de secuenciación poblacional (Sanger), aunque la relevancia de estas poblaciones minoritarias no está bien definida para los diferentes antirretrovirales y en diferentes escenarios.
En algunos estudios, las estimaciones de GSS calculadas mediante NGS ("Next Generation Sequencing") han demostrado una mayor precisión en la evaluación de las resistencias que las calculadas con la secuenciación Sanger, independientemente del algoritmo utilizado para la interpretación de las resistencias.8 Asimismo, se podría evaluar en el caso de disponer del mismo y en una evaluación conjunta con los resultados de resistencias y escore genotípico comentado anteriormente, del estudio de resistencias fenotípico -ya comentado en el capítulo 4- y del escore de sensibilidad fenotípico (phenotypic susceptibility score [PSS]). 59
El estudio de resistencias mediante la secuenciación del DNA integrado en CMSP, igualmente comentado en el capítulo 4, se podría igualmente considerar en el caso de disponer del mismo, especialmente si no se disponen de estudios de resistencias previos y/o no es posible secuenciar el RNA del virus en el FV por presencia de viremias bajas. 10
¿Cómo hay que diseñar el tratamiento de rescate? ¿número de fármacos activos, necesidad de nuevas familias?
A la hora de diseñar un tratamiento de rescate, se han planteado varias estrategias para mejorar la precisión predictiva del GSS y del PSS. Por un lado, el enfoque combinado de GSS y PSS para obtener una puntuación de susceptibilidad global (OSS) o bien, combinar la GSS/PSS para algunos fármacos junto con el historial de los tratamientos realizados.11
Un aspecto clave a la hora de optimizar una pauta de rescate optimizada es analizar la actividad esperada de los fármacos. El OSS es la suma de los fármacos totalmente activos, determinados por las pruebas fenotípica o genotípica -mediante un sistema de interpretación genotípica -habitualmente algoritmo de Standford12- en un régimen basado en todas las pruebas de resistencia disponibles, de tal forma que OSS 1 significa un fármaco totalmente activo en el OBT (Tratamiento de base optimizado, del inglés Optimized background Therapy).13 Sin embargo, en esta estrategia no tiene en cuenta la potencia del fármaco y/o la barrera a la resistencia de éste, por lo que se han planteado métodos ponderados. Algunos estudios han demostrado un claro valor de la ponderación de la potencia del fármaco para aumentar la precisión. 14
La eficacia del OBT aumenta cuando se incorporan fármacos nuevos en la terapia de rescate, lo que pone de manifiesto la importancia del uso nuevos fármacos, especialmente si son de familias nuevas, en pacientes multitratados y con alto nivel de resistencias, como FOS15 o LEN 16.
En cuanto a los fármacos a reutilizar en este escenario, se deberán valorar aquellos en los que exista actividad parcial (DRV,ETR,DOR)) y se tendrá en cuanta igualmente aquellos en los que se ha demostrado la persistencia de una actividad residual a pesar de que esté reportada resistencia genotípica completa como los análogos de nucleósidos en general 17 y el XTC en particular. 18
Recomendaciones para diseñar un TAR de rescate:
- Número de fármacos activos: el nuevo régimen debería incluir en la medida de lo posible al menos 2 fármacos totalmente activos, si uno de ellos tiene alta barrera a las resistencias, y 3 en el caso de que en el caso de que no haya un fármaco con alta berrera a las resistencias. Se define totalmente activo como: sin resistencia basada en el historial de tratamiento y/o en los genotipos disponible y con un nuevo mecanismo de acción.
- Considerar el uso de un incluir un fármaco de una familia nueva con un mecanismo de acción diferente (fostemsavir o lenacapavir, u otros fármacos que pudiera haber en el futuro) a través de su inclusión en ensayos clínicos o en programas de acceso expandido.
- Optimizar el tratamiento de base
- Valorar mantener XTC incluso en presencia de M184V. Dada la actividad residual del fármaco, la ausencia de toxicidad y de interacciones farmacológicas, y a que en se ha asociado a mejores eventos clínicos e inmunológicos. 19
- Mantener fármacos de alta barrera genética las resistencias:
- Los inhibidores de proteasa potenciados son los fármacos que han demostrado tener una mayor barrera a las resistencias y de ellos DRV/p. Si existiera alguna mutación mayor de resistencia a DRV, se recomienda utilizar DRV/r 600/100 mg BID .8
- Los INIs de segunda generación también han demostrado una alta barrera a las resistencias. El INSTI de elección para TAR de rescate es DTG, que se administrará QD o BID según el patrón de mutaciones en la integrasa y la exposición y fracaso previo o no a INI 20, BIC tiene una barrera a las resistencias similar al DTG, si bien al estar coformulado con TAF y FTC su utilización en este escenario estará condicionado al deseo de la utilización de estos fármacos
- No se recomienda añadir un solo fármaco a un régimen de TAR que está fracasando.
- No suspender el TAR.
Cambios de TAR en paciente HTE bien suprimido: simplificación, intolerancias u otros motivos
El objetivo fundamental de la optimización del TAR en un paciente multitratado en el que se ha conseguido la supresión viral es mantenerla sin poner en peligro las opciones de tratamiento futuras y manteniendo el mayor número de fármacos activos como sean posibles. En el caso de tener que modificar el TAR en un individuo HTE y con múltiples resistencias se deberá analizar con detalle el impacto de los fármacos que se vayan a modificar,analizando si las características de los fármacos que los sustituirán tienen actividad antiviral y barrera a las resistencias similares a los sustituidos. Se deberán tener en consideración todos los aspectos comentados anteriormente.
Los motivos que podrían llevar a valorar el cambio de TAR serían: reducir el número de comprimidos, frecuencia de dosificación y/o la comodidad de la posología (inyectable, ayunas…); mejorar la tolerabilidad y/o disminuir la toxicidad a corto o largo plazo; prevenir o disminuir las interacciones farmacológicas; coinfección hepatitis B; embarazo; reducir los costes.
Bibliografía:
Pelchen-Matthews A, Borges Á H, Reekie J, et al. Prevalence and Outcomes for Heavily Treatment-Experienced Individuals Living With Human Immunodeficiency Virus in a European Cohort. Journal of acquired immune deficiency syndromes (1999) 2021;87:806-17.
Human Immunodeficiency Virus-1 Infection: Developing Antiretroviral Drugs for Treatment Guidance for Industry. 2015.
Yazdanpanah Y, Fagard C, Descamps D, et al. High rate of virologic suppression with raltegravir plus etravirine and darunavir/ritonavir among treatment-experienced patients infected with multidrug-resistant HIV: results of the ANRS 139 TRIO trial. Clinical infectious diseases : an official publication of the Infectious Diseases Society of America 2009;49:1441-9.
Tassiopoulos K, Huo Y, Patel K, et al. Healthcare Transition Outcomes Among Young Adults With Perinatally Acquired Human Immunodeficiency Virus Infection in the United States. Clinical infectious diseases : an official publication of the Infectious Diseases Society of America 2020;71:133-41.
DeGruttola V, Dix L, D'Aquila R, et al. The relation between baseline HIV drug resistance and response to antiretroviral therapy: re-analysis of retrospective and prospective studies using a standardized data analysis plan. Antiviral therapy 2000;5:41-8.
Anderson JA, Jiang H, Ding X, et al. Genotypic susceptibility scores and HIV type 1 RNA responses in treatment-experienced subjects with HIV type 1 infection. AIDS Res Hum Retroviruses 2008;24:685-94.
Garcia F, Alvarez M, Fox Z, et al. Predicting antiretroviral drug resistance from the latest or the cumulative genotype. Antiviral therapy 2011;16:373-82.
Pou C, Noguera-Julian M, Pérez-Álvarez S, et al. Improved prediction of salvage antiretroviral therapy outcomes using ultrasensitive HIV-1 drug resistance testing. Clinical infectious diseases : an official publication of the Infectious Diseases Society of America 2014;59:578-88.
Hanna GJ, D'Aquila RT. Clinical use of genotypic and phenotypic drug resistance testing to monitor antiretroviral chemotherapy. Clinical infectious diseases : an official publication of the Infectious Diseases Society of America 2001;32:774-82.
Geretti AM, Blanco JL, Marcelin AG, et al. HIV DNA Sequencing to Detect Archived Antiretroviral Drug Resistance. Infectious Diseases and Therapy 2022.
Schapiro JM, Boucher CA, Kuritzkes DR, et al. Baseline CD4(+) T-cell counts and weighted background susceptibility scores strongly predict response to maraviroc regimens in treatment-experienced patients. Antiviral therapy 2011;16:395-404.
Liu TF, Shafer RW. Web resources for HIV type 1 genotypic-resistance test interpretation. Clinical infectious diseases : an official publication of the Infectious Diseases Society of America 2006;42:1608-18.
Cooper DA, Steigbigel RT, Gatell JM, et al. Subgroup and resistance analyses of raltegravir for resistant HIV-1 infection. The New England journal of medicine 2008;359:355-65.
Zazzi M, Prosperi M, Vicenti I, et al. Rules-based HIV-1 genotypic resistance interpretation systems predict 8 week and 24 week virological antiretroviral treatment outcome and benefit from drug potency weighting. The Journal of antimicrobial chemotherapy 2009;64:616-24.
Kozal M, Aberg J, Pialoux G, et al. Fostemsavir in Adults with Multidrug-Resistant HIV-1 Infection. The New England journal of medicine 2020;382:1232-43.
Segal-Maurer S, DeJesus E, Stellbrink HJ, et al. Capsid Inhibition with Lenacapavir in Multidrug-Resistant HIV-1 Infection. The New England journal of medicine 2022;386:1793-803.
Deeks SG, Hoh R, Neilands TB, et al. Interruption of treatment with individual therapeutic drug classes in adults with multidrug-resistant HIV-1 infection. The Journal of infectious diseases 2005;192:1537-44.
Campbell TB, Shulman NS, Johnson SC, et al. Antiviral activity of lamivudine in salvage therapy for multidrug-resistant HIV-1 infection. Clinical infectious diseases : an official publication of the Infectious Diseases Society of America 2005;41:236-42.
Castagna A, Danise A, Menzo S, et al. Lamivudine monotherapy in HIV-1-infected patients harbouring a lamivudine-resistant virus: a randomized pilot study (E-184V study). AIDS (London, England) 2006;20:795-803.
Eron JJ, Clotet B, Durant J, et al. Safety and efficacy of dolutegravir in treatment-experienced subjects with raltegravir-resistant HIV type 1 infection: 24-week results of the VIKING Study. The Journal of infectious diseases 2013;207:740-8.
13. CONCLUSIONES Y RECOMENDACIONES FINALES
La tasa de resistencias, tanto primarias –trasmitidas- como secundarias –adquiridas- se ha reducido de forma significativa en estos últimos años. Sin embargo, su interpretación, principalmente en el caso del paciente que presenta un fracaso virológico, sigue siendo un reto para los clínicos - en especial para los más jóvenes que no vivieron la primera década de este siglo XXI en la que éste era un escenario más frecuente-.
En estos 11 capítulos hemos querido abordar el escenario de las resistencias, no sólo desde un punto de vista técnico -revisando los principales aspectos metodológicos para la determinación de las mutaciones de resistencias y la revisión de las mutaciones que afectan la sensibilidad de los diferentes antirretrovirales-, sino también desde aspectos más conceptuales –abordando el manejo del paciente tras el fracaso virológico en diferentes situaciones de mayor complejidad: fracasos virológicos con viremias de bajo nivel, múltiples fracasos sin datos de estudios de resistencia previos-.
Como información más relevante que podríamos concluir de este documento y llevarnos a nuestras consultas, pensamos que se podrían extraer las siguientes “píldoras”:
- La disminución significativa de las resistencias transmitidas, unida a la alta barrera a las resistencias de los fármacos actualmente recomendados para el inicio del TAR (BIC,DTG), deberían no retrasar el momento de inicio del mismo hasta disponer de los resultados del ER. Disponer de esa información puede ser importante en pautas de tratamiento futuras y en cualquier caso es importante mantener la vigilancia epidemiológica de las resistencias trasmitidas a los INIs
- Los ER se deben realizar rápidamente tras la confirmación del fracaso virológico y mientras el paciente está tomando el tratamiento
- Los ER de nueva generación (NGS), capaces de detectar resistencias en poblaciones minoritarias, nos abren un nuevo e interesante “mundo” en el que nos queda mucho por aprender – i.e. aún debemos conocer cuál es el impacto de las resistencias en las poblaciones minoritarias en el fracaso virológico para los diferentes ARVs-.
- Los estudios de resistencias son una excelente herramienta de ayuda a la hora de diseñar el TAR de rescate, sin embargo, esta información siempre se debe complementar con una buena historia de los tratamientos y fracasos previos.
- Aunque en general el impacto de las diferentes mutaciones en la sensibilidad a la mayoría de los fármacos está bastante bien definido, para los fármaco que han aparecido más recientemente, TAF y los INIs –especialmente los de segunda generación, DTG y BIC-, aún es necesario recabar más información que nos ayude a mejorar su interpretación.
Esperamos que la actualización de esta guía práctica de manejo de las resistencias sea de utilidad para facilitar y optimizar el manejo clínico de las personas que viven con VIH.
Un fuerte abrazo de los redactores y coordinadores.
14. ABREVIATURAS
ABC Abacavir
ACTG Grupo de EU para estudios clínicos del SIDA ( del inglés AIDS Clinical Trials Group)
ADN Ácido desoxirribonucleico
APOBEC Enzima editora de la apolipoproteína B
ARN Ácido ribonucleico
ARVs Antirretrovirales
ATV Atazanavir
AZT Zidovudina
BIC Bictegravir
CAB Cabotegravir
CMSP Células mononucleares en sangre periférica
CR Capacidad replicativa
DBS Sangre seca en papel de filtro (del inglés Dry Blood Spot)
ddI Didanosina
d4T Estavudina
CRF Subtipos virales recombinantes circulantes (del inglés Circulating Recombinant Form)
DOR Doravirina
DRVp Darunavir potenciado
DTG Dolutegravir
EFV Efavirenz
EMA Agencia Europea del Medicamento (del inglés European Medicines Agency)
ENF Enfuvirtida o T20
EVG Elvitegravir
ER Estudios de resistencias
ETR Etravirina
FDA Organismo regulador de EU (del inglés Food and Drug Administration)
FTC Emtricitabina
FOS Fortemsavir
HTE Paciente multitratado o altamente experimentado (del inglés Heavily Treated Experienced)
IBA Ibalizumab
IN Integrasa
INIs Inhibidores de la Integrasa
IPs Inhibidores de la Proteasa
ITIAN Inhibidores de la Transcriptasa Inversa Análogos de Nucleósidos
ITINAN Inhibidores de la Transcriptasa Inversa No Análogos de Nucleósidos
LCR Líquido cefalorraquídeos
LEN Lenacapavir
LPV Lopinavir
MDR Resistentes a múltiples fármacos (del inglés Multi Drug Resistance)
MR Mutación de resistencia
MVC Maraviroc
NGS Secuenciación masiva (del inglés Next Generation Sequencing)
OBT pauta de rescate optimizada (del inglés Optimized Background Therapy)
PR Proteasa
PrEP Profilaxis pre-exposición (del inglés Pre-exposute prophylaxis)
3TC Lamivudina
r Ritonavir a dosis de potenciador
RAL Raltegravir
RPV Rilpivirina
SNC Sistema nervioso central
TAF Tenofovir alafenamida
TAMs Mutaciones para los análogos timidínicos
TAR Tratamiento antirretroviral
TDF Tenofovir disoproxil fumarato
TFV Tenofovir
TDR Transmission de Virus Resistente (del inglés Transmitted Drug Resistance)
TI Transcriptasa Inversa
URF Subtipos recombiantes únicos (del inglés Unique Recombinant Form)
VIH Virus de la Inmunodeficiencia Humana
XTC 3TC o FTC